



Sección B: Ciencias Biológicas y Ambientales
Análisis de la expresión del gen de la S-RNasa en cruzas controladas de Prunus serotina subsp. Capulí
Analysis of the expression of the S-RNase gene in controlled crosses of Prunus serotina subsp. capuli
ACI Avances en Ciencias e Ingenierías
Universidad San Francisco de Quito, Ecuador
ISSN: 1390-5384
ISSN-e: 2528-7788
Periodicidad: Bianual
vol. 15, núm. 2, 2022
Recepción: 12 Mayo 2023
Publicación: 09 Noviembre 2023
Autor de correspondencia: bioteclab@usfq.edu.ec

Resumen: El árbol de capulí (Prunus serotina subsp.capul) es nativo de América del Norte, pero se encuentra ampliamente distribuido en zonas frías y templadas de Sudamérica, especialmente en Ecuador. Su fruto, una pequeña baya, es de gran interés comercial debido a sus propiedades terapéuticas como antioxidantes, que están asociadas al tratamiento de enfermedades cardiovasculares e inflamatorias. Tanto los productores nacionales como los pueblos ancestrales aprovechan este recurso para elaborar mermeladas y bebidas fermentadas y/o espirituosas. Aunque no se cultivan de forma agrícola en Ecuador, resulta intrigante estudiar sus mecanismos de reproducción para aplicarlos en programas de agricultura y reforestación. En especies como P. serotina es interesante entender el mecanismo GSI (gametophytic self-incompatibilit.) que ha evolucionado en flores hermafroditas para evitar la endogamia y, de esa manera, limitar la producción de frutos. El GSI está regulado por el Locus S, que consiste en dos genes cruciales: el gen S-RNasa (expresado en el pistilo) y el gen SFB (expresado en el polen). La interacción entre estos dos genes activa o desactiva ciertas proteínas que determinan la compatibilidad o incompatibilidad de una determinada cruza, funcionando como un complejo molecular similar a una llave y una cerradura. Gracias a este sistema, se evita que las cruzas entre individuos genéticamente emparentados generen frutos, ya que se trunca el crecimiento de los tubos polínicos, impidiendo que lleguen al ovario y fecunden el óvulo. Con el objetivo de estudiar la expresión del gen S-RNasa en cruzas compatibles e incompatibles del capulí, se llevaron a cabo polinizaciones controladas y se extrajo el ARN para realizar una RT-qPCR. Los análisis estadísticos de los valores C. obtenidos de las qPCR revelaron que no existe una diferencia significativa en la expresión del gen S-RNasa entre las cruzas compatibles e incompatibles, mediante un ANOVA (p = 0,0632). Para complementar estos hallazgos, se realizó un análisis del desarrollo de los tubos polínicos en las distintas cruzas, lo que permitió una mejor comprensión de su relación con la expresión del gen S-RNasa. Se observó que, en las cruzas incompatibles, el 50 % de tubos polínicos tuvieron un crecimiento hasta el estilo. Estos resultados sugieren que, en el caso del capulí, podría ocurrir una supresión del sistema GSI, lo que provocaría que cruzas consideradas como incompatibles actúen como compatibles. Este estudio proporciona valiosa información sobre los mecanismos de reproducción del capulí y puede tener implicaciones importantes para su cultivo y conservación. Sin embargo, se necesita una investigación más profunda para comprender completamente este fenómeno y su potencial impacto en la agricultura y reforestación. Se espera que estos conocimientos contribuyan al desarrollo sostenible de esta especie y a su utilización responsable en beneficio de las comunidades locales y el ecosistema en general.
Palabras clave: capulí, Prunus serotina subsp capuli, Sistema de Incompatibilidad Gametofítica (GSI), S-RNasa, RT-qPCR.
Abstract: The capuli tree (Prunus serotina subsp.capuli) is native to North America but is widely distributed in cold and temperate regions of South America, especially in Ecuador. It’s fruit, a small berry, is of great commercial interest due to its therapeutic properties as antioxidants, which are associated with the treatment of cardiovascular and inflammatory diseases. Both national producers and indigenous communities harness this resource to make jams and fermented beverages. Although is not cultivated agriculturally in Ecuador, it is intriguing to study its reproduction mechanisms for application in agriculture and reforestation programs. In species like P. serotina, it is interesting to understand the mechanism of GSI (gametophytic self-incompatibility) that has evolved in hermaphroditic flowers to prevent inbreeding and thus limit fruit production. GSI is regulated by the S locus, which consists of two crucial genes: the S-RNase gene (expressed in the pistil) and the SFB gene (expressed in the pollen). The interaction between these two genes activates or deactivates certain proteins that determine the compatibility or incompatibility of a particular cross, functioning as a molecular complex similar to a lock and key system. Because of this system, crosses between genetically related individuals are prevented from producing fruit, as the growth of pollen tubes is truncated, preventing them from reaching the ovary and fertilizing the ovule. With the aim of studying the expression of the S-RNase gene in compatible and incompatible crosses of the capuli, controlled pollinations were carried out, and RNA was extracted for RT-qPCR analysis. Statistical analysis of the C. values obtained from the qPCR revealed no significant difference in the expression of the S-RNase gene between compatible and incompatible crosses, as determined by ANOVA (p = 0,0632). To complement these findings, an analysis of pollen tube development in the different crosses was performed, allowing for a better understanding of its relationship with the expression of the S-RNase gene. It was observed that in incompatible crosses, 50% of pollen tubes exhibited growth up to the style. These results suggest that in the case of capuli, suppression of the GSI system could occur, causing crosses considered incompatible to act as compatible. This study provides valuable information about the reproduction mechanisms of the capuli and may have significant implications for its cultivation and conservation. However, further research is needed to fully understand this phenomenon and its potential impact on agriculture and reforestation. It is hoped that this knowledge will contribute to the sustainable development of this species and its responsible use for the benefit of local communities and the ecosystem as a whole.
Keywords: capuli, Prunus serotina subsp. capuli, Gametophytic Incompatibility System (GSI), S-RNase, RT-qPCR.
Introducción
Prunus serotina subsp. capuli (Cav. ex Spreng.) McVaugh pertenece a la familia Rosaceae. Se lo conoce como capulí y es originario de Norte América, pero su distribución llega hasta América del Sur, sobre todo en la región andina de Ecuador, en zonas frías y templadas sobre los 2000 metros a nivel del mar [1]. En cada racimo, florecen de 10 a 20 flores blancas, de aproximadamente 1 cm, entre los meses de agosto y octubre. De estas se obtiene una baya verde que madura a finales de noviembre, adquiriendo tonalidades rojizas, marrones y negras [2].
En el Ecuador, tanto el fruto y las hojas, como su madera, tienen un alto potencial comercial [3-5], sin embargo, no se han establecido cultivos agrícolas para potenciar su producción [6]. Se ha reportado que el fruto cuenta con propiedades antioxidantes y antiiflamatorias que ayudan a combatir diferentes enfermedades tanto cardiovasculares como neurodegenerativas [7-9]. Los pueblos ancestrales ecuatorianos consideran al capulí un fruto tradicional para la preparación de la sagrada bebida `jucho ´, además de que las comunidades andinas lo utilizan como fuente de ingresos para elaboración de mermeladas [10,11].
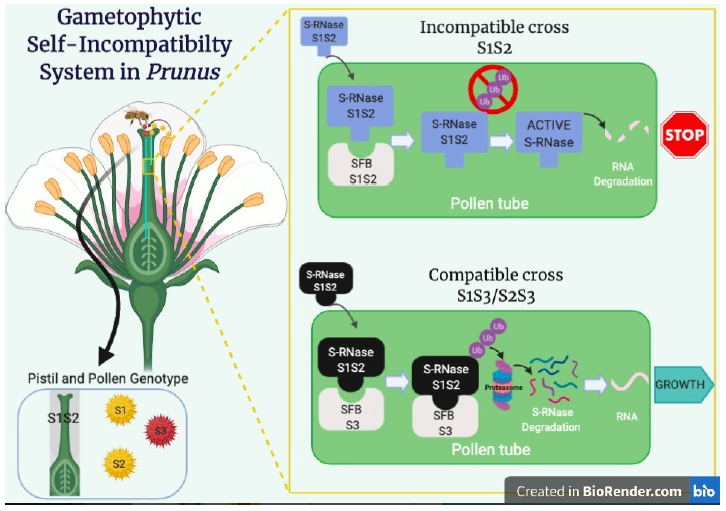
Al igual que en otras especies del género Prunus, su flor es hermafrodita, es decir que tiene la parte reproductora femenina y masculina, lo que le ha llevado a desarrollar el Sistema de Incompatibilidad Gametofítica también llamado GSI (gametophytic self-incompatibility), con el fin de conservar la diversidad genética de la especie. Este sistema es controlado por el locus S, compuesto por el gen S-RNasa expresado en el pistilo, y el gen SFB expresado en el polen [12]. Ambos genes se encargan de producir proteínas específicas que al interactuar entre sí, determinan la compatibilidad o incompatibilidad de las cruzas [13]. El gen de la S-RNasa codifica proteínas específicas para el reconocimiento del polen y para activar las ribonucleasas que hidrolizan las cadenas de ARN [12,14,15].
Este sistema funciona debido a la interacción de los haplotipos (variación de un mismo gen) presentes tanto en el pistilo como en el polen, conocidos como haplotipos S (Figura 1). Por lo tanto, para considerar una cruza como incompatible, el haplotipo S (S1) del polen debe corresponder con alguno de los haplotipos S (S1 o S2) del pistilo. Si ambos coinciden con el haplotipo S1, el mecanismo se activará, provocando que la S-RNasa no pueda formar el complejo molecular con la proteína SFB, y la S-RNasa permanecerá activa y degradará al ARN de los tubos polínicos. De esta manera, se detendrá el crecimiento, y el desarrollo del fruto se verá afectado [16,15,17]. En cambio, al observar una cruza compatible, el haplotipo (S1) del polen, diferirá del haplotipo (S2) del pistilo, provocando que la S-RNasa encaje con el SFB formando el complejo molecular. Esto desencadena que las proteínas ubiquitinadas degraden a la S-RNasa, y que el ARN de los tubos polínicos no se vea afectado [18].
El objetivo de esta investigación fue cuantificar y comparar la expresión del gen de la S-RNasa en diferentes cruzas controladas del capulí y analizar el crecimiento de tubos polínicos para esclarecer su función en el sistema GSI.
Materiales y métodos
Selección de muestras
Los árboles utilizados en esta investigación se ubican en la hacienda “San Felipe” en Cayambe (N 00,04049 W 078,15900). Los árboles de capulí se encuentran sembrados junto al otro en una línea recta en una zona de la hacienda que tiene otros árboles frutales. Los árboles de capulí fueron numerados del 1 al 22 considerando su ubicación de sur a norte. Los árboles utilizados en el estudio fueron: 1, 14, 15, 17 y 22 (Tabla 1).
| Tipo de cruza | Árboles del estudio | ||
| Árbol receptor (pistilo) | Árbol donador (polen) | Alelos [30] | |
| Compatible | 1 | 17 | 1: S8 S19 17: S4 S1 |
| Compatible | 15 | 14 | 15: S10 S6 14: S20 S21 |
| Incompatible | 17 | 17 | S4 S1 |
| Incompatible | 17 | 22 | S4 S1 |
Los alelos de la S-RNasa presentes en cada árbol se determinaron mediante marcadores moleculares CAPS diseñados para la especie [19]. Con esta información se determinaron cruzas compatibles e incompatibles [20]. Para las cruzas compatibles se utilizaron individuos con diferentes alelos, mientras que en las cruzas incompatibles se usaron individuos con los mismos alelos. Para mantener la homogeneidad de muestras, se consideraron 2 cruzas compatibles (1 × 17 y 15 × 14) y 2 incompatibles (17 × 22 y 17 × 17), donde la cruza 17 × 17 fue una autopolinización, es decir que se polinizó con granos de polen del mismo árbol [20].
Polinizaciones controladas
Para cada cruza, se extrajo polen de 50 flores con botones cerrados en estadío G [2]. Se usaron 120 flores en el estadío F y G [2] que fueron emasculadas (extracción de las anteras) para realizar las polinizaciones. Se verificó que la viabilidad del polen fuera superior al 80% con una tinción de Acetocarmín [21, 20]. Las 120 flores emasculadas se separaron: 60 para polinizar y 60 para el control (no polinizadas). A las 48 horas, el material vegetal fue recuperado, 100 pistilos (50 polinizados/50 control) se utilizaron para el análisis de la expresión de la S-RNasa y 20 pistilos (10 polinizados/10 control) para teñir con azul de anilina y ver mediante microscopía de fluorescencia el crecimiento de los tubos polínicos a lo largo del pistilo [20].
Análisis de la expresión de la S-RNasa mediante qPCR
Se extrajo ARN de los pistilos recoletados después de las 48 horas de la polinización usando el protocolo de Gambino et al. [22] y Gómez et al. [23], agregando 2,6 µL de Glycoblue para teñir al pellet en el paso de precipitación con isopropanol. El ADN complementario se obtuvo con la metodología de Pazmiño [24].
Para analizar la expresión del gen en tiempo real, se realizó un RT-qPCR, utilizando el gen housekeeping RPII, reportado como el más estable en la literatura [25], debido a su mínima variación de expresión en los diferentes tratamientos. Se debe contar con un gen housekeeping dentro de los experimentos para incrementar la confiabilidad de los resultados ya que sirve como base para calcular la expresión del gen de interés (S-RNasa). Las secuencias de los primers usados del gen de la S-RNasa y del gen RPII se tomaron de Gómez [25](Tabla 2). La PCR en tiempo real fue realizada en el termociclador CFX96 TOUCH™ Bio-Rad, usando el protocolo de Erazo [26]. El protocolo consistió en una desnaturalización inicial a 95°C durante 10 min, luego durante 50 ciclos, se realizó una desnaturalización a 95 °C por 15 s, una hibridación a 55 °C por 30 s y una extensión a 72 °C por 30 s.
| Gen | Especie referencial | Primer | Referencia | |
| Forward | Reverse | |||
| S-RNasa | Prunus dulcis | TGCACTGGGTCGCAATTTAA | TGTTCCACTCGTCTTCCCAA | [25] |
| RPII | Prunus dulcis | TGAAGCATACACCTATGATGATGAAG | CTTTGACAGCACCAGTAGATTCC | [25] |
Análisis estadísticos
En el RT-qPCR se realizaron 3 réplicas técnicas para obtener los valores Ct, los cuales indican la expresión del gen de la S-RNasa. Estos valores fueron analizados por el método de cuantificación relativa 2^-ΔΔ Ct. Posteriormente, se normalizaron los datos mediante el software Minitab, y se realizaron pruebas estadísticas para determinar si la expresión diferencial era significativa [27]. Mediante un ANOVA de un factor en el programa R se comparó las medias entre las muestras polinizadas de las cruzas compatibles e incompatibles y obtener el valor p.
Análisis del crecimiento de los tubos polínicos mediante microscopia de fluorescencia
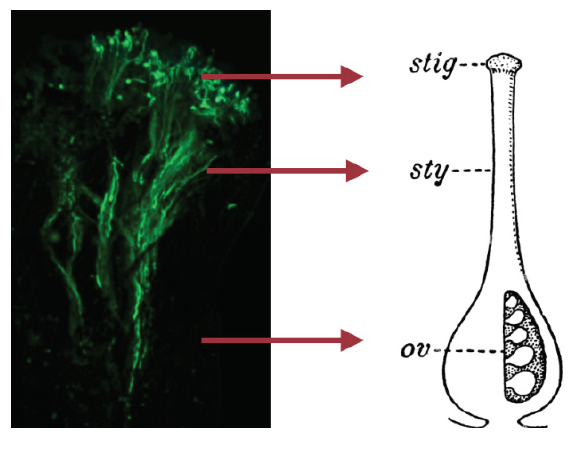
Para verificar los efectos de la expresión del gen a lo largo del pistilo, se debe entender la fisiología de los pistilos, que se divide principalmente en 3 áreas: estigma (stig), estilo (sty) y ovario (ov) (Figura 2).
Los pistilos se tiñeron con azul de anilina usando el protocolo de Baquero [20]. Posteriormente, se los observó bajo un microscopio de fluorescencia. Este tinte se une con un polisacárido presente en las membranas vegetales conocido como calosa, que se halla tanto en las paredes de los tubos polínicos como alrededor de los granos de polen, provocando una coloración azul verdosa cuando se expone al material a una luz especial azul o ultravioleta [28].
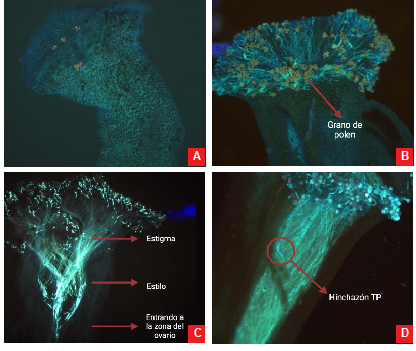
Para analizar visualmente el crecimiento de los tubos polínicos, primero se tomó en cuenta la presencia o ausencia de polen en el estigma (Figura 3A-3B). Se observó la presencia de polen (en muestras polinizadas), esto indicó una polinización exitosa (Figura 3B). Segundo, se contó el número de tubos polínicos que se encontraban en cada sección del pistilo (estigma, estilo y ovario) (Figura 3C). Por último, se revisó si es que las puntas de los tubos polínicos presentaban una hinchazón acompañada del cese de la fluorescencia, ya que esto indica un truncamiento en el crecimiento de los tubos polínicos (Figura 3D).
Resultados
Expresión de la S-RNasa
A partir de los valores Ct obtenidos de la RT-qPCR, se realizó una comparación de la expresión de la S-RNasa de las muestras polinizadas entre cruzas compatibles e incompatibles, en términos del valor Ct (Figura 4).
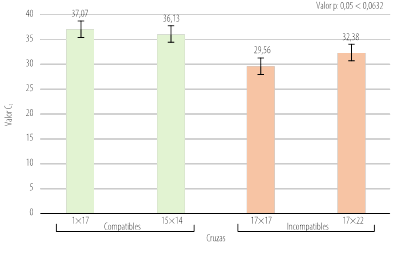
Se observan las medias de los valores Ct de cada una de las cruzas con el valor p correspondiente, obtenido al comparar las medias de expresión entre los pistilos polinizados de las cruzas compatibles e incompatibles mediante un ANOVA de un factor que indica que entre los pistilos polinizados de las cruzas compatibles e incompatibles no se muestra una diferencia estadística significativa.
Las cruzas compatibles tuvieron una media de 30,97 y las incompatibles tuvieron una media de 36,60. Al comparar ambos valores de la expresión de la S-RNasa de las cruzas compatibles e incompatibles, a través de un ANOVA de un factor, se obtuvo un valor p 0,05 < 0,0632, lo que indica que no existe una diferencia significativa en la expresión de la S-RNasa, datos detallados en la Tabla 3 y complementados con la Figura 4.
| Tipo de cruza | Media | Valor p |
| Compatible | 30,97 | 0,0632 |
| Incompatible | 36,60 |
Crecimiento de tubos polínicos en cada cruza
En cuanto a las cruzas compatibles, en la cruza 1×17, el 18,2 % de tubos polínicos creció hasta el estigma, y el 45,5 % llegó hasta el estilo, mientras que no se obtuvo visualización de tubos polínicos hasta el ovario. En los pistilos control, el porcentaje de tubos polínicos que llegaron al estigma fue mínimo (9,1 %). En la cruza 15×14 no se pudo visualizar ni la presencia de polen ni el desarrollo de tubos polínicos en ninguna de las secciones del pistilo polinizado, al igual que en las muestras control lo que indica que no se logró una polinización exitosa (Tabla 4).
| Cruzas | ||||||||
| Compatibles | Incompatibles | |||||||
| 1×17 | 15×14 | 17×17 | 17×22 | |||||
| Desarrollo de TP | Polinizadas | Control | Polinizadas | Control | Polinizadas | Control | Polinizadas | Control |
| (1/3) estigma | 18,2 % | 9,1 % | - | - | 38,89 % | 10,53 % | 23,09 % | 33,33 % |
| (2/3) estilo | 45,5 % | 0 % | - | - | 50 % | 68,42% | 46,15 % | 66,67 % |
| (3/3) ovario | 0 % | 0 % | - | - | 11,11 % | 0 % | 7,69 % | 0 % |
| Sin polen/ sin desarrollo | 36,3 % | 90,9 % | 100 % | 100 % | 0 % | 21,05 % | 23,07 % | 0 % |
Con respecto a las cruzas incompatibles, en la cruza 17 × 17 se observó un porcentaje alto de crecimiento de tubos polínicos, donde el 38,89 % alcanzó el estigma, mientras que el 50 % se estancó en el estilo. Solamente el 11 % de los tubos polínicos se desarrollaron hasta el ovario. En los pistilos control también se reportó un alto crecimiento de tubos polínicos del 68,42 % que se desarrollaron únicamente hasta el estilo. En la cruza 17 × 22 se registró el 23,09 % de tubos polínicos que crecieron hasta el estigma, el 46,15 % se desarrolló hasta el estilo, y solamente el 7,69 % llegó hasta la tercera parte del pistilo que corresponde a la zona del ovario. En las muestras control el 66,67 % de tubos polínicos se desarrollaron hasta el estilo (Tabla 4).
Discusión
Comparación de la expresión de la S-RNasa en cruzas compatibles e incompatibles
Como se ha mencionado, uno de los objetivos de esta investigación fue estudiar la expresión del gen de la S-RNasa y cómo su expresión está relacionada con el crecimiento de tubos polínicos a lo largo del estilo. Los datos de la Tabla 3 indican que las cruzas incompatibles, es decir, aquellas que son genéticamente emparentadas (presentan los mismos alelos), muestran una mayor expresión del gen de la S-RNasa en comparación a las cruzas compatibles. Se espera que la expresión de este gen sea mayor en cruzas incompatibles ya que este se encarga de codificar proteínas que degradan el ARN, lo que genera que se detenga el crecimiento de los tubos polínicos dentro del pistilo, y de esta manera evitar la fecundación, lo que ha sido ampliamente reportado en la literatura [12-15].
Desarrollo de tubos polínicos en las diferentes cruzas
Con respecto a las cruzas compatibles, en la cruza 15 × 14, en los pistilos polinizados no se observó crecimiento de tubos polínicos en ninguna de las secciones (estigma, estilo, ovario) y se confirmó que tampoco había polen en el estigma. Esto indica que algunos factores influyeron posiblemente en la polinización como la receptividad del pistilo o la temperatura del ambiente. La receptividad se refiere a la capacidad del estigma para beneficiar la posible germinación de polen [33]. Por lo tanto, se debe considerar el estado de recolección del pistilo, ya que según Ramírez & Davenport [2], cuando las flores se encuentran en el estado recomendado (F o G), próximas a abrirse, secretan sustancias que ayudan a la germinación del polen hallándose más receptivas [33]. La receptividad también se puede ver afectada por la temperatura. De hecho, para que una fertilización sea exitosa es importante considerar la temperatura en la que se mantiene el pistilo [34]. En este caso, la temperatura del lugar (Cayambe) donde se recolectaron las flores no supera los 20 °C, mientras que, en donde se realizaron los procesos de polinización y extracción (Cumbayá), la temperatura llegaba hasta los 25 °C . Se conoce que en diversos cultivares de Prunus cuando son expuestos a altas temperaturas, aproximadamente 23-25 °C, puede reducirse la efectividad de la polinización, influyendo tanto en la cantidad de granos de polen que se adhieren al estigma, como en la velocidad de crecimiento de tubos polínicos [35].
De acuerdo con los resultados obtenidos, en las cruzas incompatibles es posible suponer que el sistema GSI ha sufrido algún rompimiento, ya que se observa un alto porcentaje de los tubos polínicos que llegaron hasta el ovario, específicamente en aquellas cruzas del árbol 17 (17 × 17 y 17 × 22) (Tabla 4). En los experimentos realizados por Gordillo et al. [30], se observa un comportamiento similar en los pistilos polinizados de las mismas cruzas (17 × 17 y 17 × 22), donde se reporta un alto porcentaje de desarrollo de tubos polínicos que llegan hasta el estilo y algunos, inclusive, hasta el ovario.
Implicación de un posible rompimiento del GSI
Es esencial destacar que los resultados obtenidos en este estudio han contribuido significativamente a nuestra comprensión de la influencia del gen de la S-RNasa y han sentado las bases para futuras investigaciones en el campo de la incompatibilidad gametofítica (GSI) en Prunus serotina subsp.capuli [20, 30]. Sin embargo, para obtener una visión completa y detallada de este sistema, es necesario llevar a cabo investigaciones que aborden otros aspectos importantes, como la expresión y la influencia del gen SFB [38].
En otras especies de Prunus pueden ocurrir mutaciones que implican la pérdida de funcionalidad genética en el gen de la S-RNasa y en el gen SFB [31, 32, 39, 40, 41]. En estas circunstancias, el sistema de incompatibilidad gametofítica (GSI) no se activa, lo que implica que el crecimiento de los tubos polínicos pueda alcanzar la zona del ovario y, potencialmente, fecundar el óvulo y formar frutos. Esta ruptura en el GSI se ha reportado con mayor frecuencia en el gen SFB y se ha observado en especies Prunus como P. avium, P. armeniaca, P. dulcis, P. pyrifolia y P. saliciana [42-47].
La investigación de las mutaciones que puedan afectar a la funcionalidad del GSI en el capulí, no solo tiene importancia biológica, sino que también tiene implicaciones prácticas relevantes en el ámbito agrícola. Esta disrupción en el sistema se considera una ventaja prometedora, ya que puede mejorar la eficiencia de los cultivos y conducir a una mayor producción de frutos. Esto brinda una valiosa oportunidad económica para los agricultores locales y puede crear una base sólida para tomar decisiones informadas sobre la selección de variedades de capulí que tengan una producción eficiente [46-50]. Tener mayor información de procesos biológicos como es el GSI, contribuye para que los agricultores puedan optimizar sus prácticas de cultivo y, al mismo tiempo, preserven la variabilidad genética de la especie, que es esencial para combatir enfermedades, plagas y condiciones climáticas cambiantes que pueden afectar el éxito de la producción agrícola [49,50].
Agradecimientos
Las autoras agradecen a los miembros del Laboratorio de Biotecnología Vegetal por su apoyo en la realización de esta investigación. También agradecen a José Tobar por el acceso a los árboles en la hacienda “San Felipe”.
Referencias
[1] León, J. (2000). Fundamentos Botánicos de los Cultivos Tropicales. Instituto Interamericano de Cooperación para la Agricultura (IICA).
[2] Ramírez, F. y Davenport, T. (2016). The phenology of the capuli cherry [Prunus serotina subsp. capuli (Cav.) McVaugh] characterized by the BBCH scale, landmark stages and implications for urban forestry in Bogotá, Colombia. Urban Forestry & Urban Greening, 19, 202-211. doi: https://doi.org/10.1016/j.ufug.2016.06.028
[3 Málaga, R. et. al. (2009). Caracterización y evaluación de los recursos naturales de la microcuenca cunyatupe. Revista del Instituto de Investigación de la Facultad de Ingeniería Geológica, Minera, Metalúrgica y Geográfica, 12(23), 12-20. https://dialnet.unirioja.es/servlet/articulo?codigo=8135799
[4] Baños, K. (2017). Identificación y descripción de las características anatómicas de la madera de Prunus serotina (Capulí) procedente de tres provincias: Chimborazo, Tungurahua y Cotopaxi. Escuela Superior Politécnica de Chimborazo. http://dspace.espoch.edu.ec/handle/123456789/6683
[5] Cueva, C. (2019). Etnobotánica de las plantas medicinales del caserío Laguna San Nicolas. Universidad Nacional de Cajamarca
[6] Monteros, A., Tapia, C. y Borja, E. J. (2013). Colecta y caracterización morfoagronómica in situ y molecular de capuli (Prunus serotina Ehrh) del banco nacional de germoplasma del INIAP-Ecuador. INIAP, Estación Experimental Santa Catalina. http://repositorio.iniap.gob.ec/handle/41000/928
[7] Zavala, D. et. al. (2006). Efecto citotóxico de Physalis peruviana (capulí) en cáncer de colon y leucemia mieloide crónica. Anales de la Facultad de Medicina, 67(4), 283-289. http://www.scielo.org.pe/scielo.php?script=sci_arttext&pid=S1025-55832006000400002
[8] Torres, R. y Teves, F. (2011). Estudio comparativo de la actividad antioxidante in vitro de los extractos antociánicos y caracterización de las antocianidinas en los frutos de las especies vegetales Prunus serótina (capuli) [Tesis, Universidad Nacional de San Antonio de Cusco]. Repositorio Institucional Universidad Nacional de San Antonio de Cusco. http://hdl.handle.net/20.500.12918/1061
[9] Ruiz, S. (2018). Características farmacognósticas y cuantificación espectrofotométrica de antocianinas totales del fruto de Prunus serotina subsp. capuli (Cav.) McVaugh (Rosaceae) “capulí”. Arnaldoa, 25(3), 961-980. http://www.scielo.org.pe/scielo.php?script=sci_arttext&pid=S2413-32992018000300009
[10] Ruales Estupiñán, C. (2007). Estudios para la recuperación de la flora nativa en el valle de Tumbaco-Distrito Metropolitano de Quito: inventario florístico y ensayo de propagación vegetativa [Tesis de Máster, Universidad San Francisco de Quito USFQ]. Repositorio Digital USFQ. http://repositorio.usfq.edu.ec/handle/23000/886
[11] Acosta, A. (2019). Caracterización carpológica de la especie de uso alimenticio Prunus serotina Kunth 1879 en la Zona Central de los Andes del Ecuador [Tesis de Pregrado, Escuela Superior Politécnica de Chimborazo]. DSpace ESPOCH. http://dspace.espoch.edu.ec/handle/123456789/12342
[12] Roalson, E. H. y McCubbin, A. G. (2003).S-RNases and sexual incompatibility: structure, functions, and evolutionary perspectives. Molecular phylogenetics and evolution, 29(3), 490-506. doi: https://doi.org/10.1016/S1055-7903(03)00195-7
[13] De Nettancourt, D. (2001). Incompatibility and incongruity in wild and cultivated plants. Springer Berlin, Heidelberg. doi: https://doi.org/10.1007/978-3-662-04502-2
[14] Yamane, H. y Tao, R. (2009). Molecular basis of self-(in) compatibility and current status of S-genotyping in Rosaceous fruit trees. Journal of the Japanese Society for Horticultural Science, 78(2), 137-157. doi: https://doi.org/10.2503/jjshs1.78.137
[15] Tao, R. y Iezzoni, A. F. (2010). The S-RNase-based gametophytic self-incompatibility system in Prunus exhibits distinct genetic and molecular features. Scientia Horticulturae, 124(4), 423-433. https://doi.org/10.1016/j.scienta.2010.01.025
[16] Hua, Z. H., Fields, A. y Kao, T. H. (2008). Biochemical models for S-RNase-based self-incompatibility. Molecular Plant, 1(4), 575-585. doi: https://doi.org/10.1093/mp/ssn032
[17] Wu, J., Gu, C., Khan, M. A., Wu, J., Gao, Y., Wang, C. y Zhang, S. (2013). Molecular determinants and mechanisms of gametophytic self-incompatibility in fruit trees of Rosaceae. Critical Reviews in Plant Sciences, 32(1), 53-68. doi: https://doi.org/10.1080/07352689.2012.715986
[18] McClure, B. (2009). Darwin’s foundation for investigating self-incompatibility and the progress toward a physiological model for S-RNase-based SI. Journal of experimental botany, 60(4), 1069-1081. doi: https://doi.org/10.1093/jxb/erp024
[19] Correa, L. (2018). Caracterización molecular y diseño de marcadores moleculares CAPS para el gen de la S-RNasa en Prunus serotina subsp. capuli [Tesis de Pregrado, Universidad San Francisco de Quito USFQ]. Repositorio Digital USFQ. http://repositorio.usfq.edu.ec/handle/23000/7363
[20] Baquero Méndez, V. Y. (2018). Estudio preliminar fenotípico de la incompatibilidad gametofítica en capulí (Prunus serotina subsp. capuli) [Tesis de Pregrado, Universidad San Francisco de Quito USFQ]. Repositorio Digital USFQ. http://repositorio.usfq.edu.ec/handle/23000/7782
[21] Asma, B. M. (2008). Determination of pollen viability, germination ratios and morphology of eight apricot genotypes. African Journal of Biotechnology, 7(23). https://www.ajol.info/index.php/ajb/article/view/59562
[22] Gambino, G., Perrone, I. y Gribaudo, I. (2008). A rapid and effective method for RNA extraction from different tissues of grapevine and other woody plants. Phytochemical analysis, 19(6), 520-525. doi: https://doi.org/10.1002/pca.1078
[23] Gómez, E. M., Dicenta, F., Martínez-García, P. J. y Ortega, E. (2015). iTRAQ-based quantitative proteomic analysis of pistils and anthers from self-incompatible and self-compatible almonds with the S f haplotype. Molecular Breeding, 35(5), 1-14. https://link.springer.com/article/10.1007/s11032-015-0314-5
[24] Pazmiño Cajiao, M. A. (2018). Evaluación de crecimiento y expresión de genes relacionados a las rutas metabólicas del ácido jasmónico y ácido salicílico después de la aplicación de glifosato en Arabidopsis thaliana [Tesis de Pregrado, Universidad San Francisco de Quito USFQ]. Repositorio Digital USFQ. http://repositorio.usfq.edu.ec/handle/23000/7533
[25] Gómez, E. M., Buti, M., Sargent, D. J., Dicenta, F. y Ortega, E. (2019). Transcriptomic analysis of pollen-pistil interactions in almond (Prunus dulcis) identifies candidate genes for components of gametophytic self-incompatibility. Tree Genetics & Genomes, 15(4), 1-13. https://link.springer.com/article/10.1007/s11295-019-1360-7
[26] Erazo García, M. P. (2019). Determinación de la producción de alcaloides y análisis de expresión de genes de defensa inducidos por metil jasmonato en semillas de chocho (Lupinus mutabilis sweet) [Tesis de Pregrado, Universidad San Francisco de Quito USFQ]. Repositorio Digital USFQ. http://repositorio.usfq.edu.ec/handle/23000/7800
[27] Molina Arias, M. (2017). ¿Qué significa realmente el valor de p? Pediatría Atención Primaria, 19(76), 377-381.
[28] Radičević, S., Cerović, R., Nikolić, D. y Đorđević, M. (2016). The effect of genotype and temperature on pollen tube growth and fertilization in sweet cherry (Prunus avium L.). Euphytica, 209(1), 121-136. doi: https://doi.org/10.1007/s10681-016-1645-y
[29] García-Valencia, L. E., Bravo-Alberto, C. E. y Cruz-García, F. (2013). Evitando el incesto en las plantas: control genético y bioquímico. TIP Revista Especializada en Ciencias Químico-Biológicas, 16(1), 57-65. https://www.medigraphic.com/cgi-bin/new/resumen.cgi?IDARTICULO=42710
[30] Gordillo-Romero, M., Correa-Baus, L., Baquero-Méndez, V., de Lourdes Torres, M., Vintimilla, C., Tobar, J. y Torres, A. F. (2020). Gametophytic self-incompatibility in Andean capuli (Prunus serotina subsp. capuli): allelic diversity at the S-RNase locus influences normal pollen-tube formation during fertilization. PeerJ, 8, e9597. doi: https://doi.org/10.7717/peerj.9597
[31] Yamane, H., Tao, R., Sugiura, A., Hauck, N. R. y Iezzoni, A. F. (2001). Identification and characterization of S-RNases in tetraploid sour cherry (Prunus cerasus). Journal of the American Society for Horticultural Science, 126(6), 661-667. doi: https://doi.org/10.21273/JASHS.126.6.661
[32] Hauck, N. R., Yamane, H., Tao, R. y Iezzoni, A. F. (2002).Self-compatibility and incompatibility in tetraploid sour cherry (Prunus cerasus L.). Sexual Plant Reproduction, 15(1), 39-46. doi: https://doi.org/10.1007/s00497-002-0136-6
[33] Yi, W., Law, S. E., McCoy, D. y Wetzstein, H. Y. (2006). Stigma development and receptivity in almond (Prunus dulcis). Annals of Botany, 97(1), 57-63. doi: https://doi.org/10.1093/aob/mcj013
[34] Hedhly, A. (2003). Efecto de la temperatura sobre la fase reproductiva en cerezo (Prunus avium L.) [Tesis doctoral, Universidad de Lleida]. Digital CSIC. http://hdl.handle.net/10261/128750
[35] DeCeault, M. T. y Polito, V. S. (2008). High temperatures during bloom can inhibit pollen germination and tube growth, and adversely affect fruit set in the prunus domestica cultvars’improved french’and’muir beauty’. Acta Horticulturae 874(874), 163-168. https://www.researchgate.net/publication/284250781_High_temperatures_during_bloom_can_inhibit_pollen_germination_and_tube_growth_and_adversely_affect_fruit_set_in_the_Prunus_domestica_cultvars_’Improved_French’_and_’Muir_Beauty’
[36] Orlando Marchesano, B. M., Chiozzotto, R., Baccichet, I., Bassi, D. y Cirilli, M. (2022). Development of an HRMA-Based Marker Assisted Selection (MAS) Approach for Cost-Effective Genotyping of S and M Loci Controlling Self-Compatibility in Apricot (Prunus armeniaca L.). Genes, 13(3), 548. doi: https://doi.org/10.3390/genes13030548
[37] Dongariyal, A., Dimri, D. C., Kumar, P., Choudhary, A., Jat, P. K., Basile, B. y Singh, A. (2022). Pollen-Pistil Interaction in Response to Pollination Variants in Subtropical Japanese Plum (Prunus salicina Lindl.) Varieties. Plants, 11(22), 3081. doi: https://doi.org/10.3390/plants11223081
[38] Sullivan, J. A., Shirasu, K. y Deng, X. W. (2003). The diverse roles of ubiquitin and the 26S proteasome in the life of plants. Nature Reviews Genetics, 4, 948-958. doi: https://doi.org/10.1038/nrg1228
[39] Tsukamoto, T., Ando, T., Kokubun, H., Watanabe, H., Sato, T., Masada, M. y Kao, T. H. (2003). Breakdown of self-incompatibility in a natural population of Petunia axillaris caused by a modifier locus that suppresses the expression of an S-RNase gene. Sexual Plant Reproduction, 15(5), 255-263. doi: https://doi.org/10.1007/s00497-002-0161-5
[40] Hancock, C., Kent, L. y McClure, B. (2005). The stylar 120 kDa glycoprotein is required for S-specific pollen rejection in Nicotiana. The Plant Journal, 43, 716-723. doi: https://doi.org/10.1111/j.1365-313X.2005.02490.x
[41] Li, Y., Wu, J., Wu, C., Yu, J., Liu, C., Fan, W. y Li, W. (2020). A mutation near the active site of S-RNase causes self-compatibility in S-RNase-based self-incompatible plants. Plant molecular biology, 103(1), 129-139. doi: https://doi.org/10.1007/s11103-020-00979-z
[42] Cardozo, T. y Pagano, M. (2004). The SCF ubiquitin ligase: insights into a molecular machine. Nature Rev. Mol. Cell Biol. 5, 739-751. doi: https://doi.org/10.1038/nrm1471
[43] Sonneveld, T., Tobutt, K. R., Vaughan, S. P. y Robbins, T. P. (2005). Loss of pollen-S function in two self-compatible selections of Prunus avium is associated with deletion/mutation of an S haplotype–specific F-Box gene. The Plant Cell, 17(1), 37-51. doi: https://doi.org/10.1105/tpc.104.026963
[44] Ahmad, M. H., Rao, M. J., Hu, J., Xu, Q., Liu, C., Cao, Z. y Chai, L. (2022). Systems and breakdown of self-incompatibility. Critical Reviews in Plant Sciences, 41(3), 209-239. doi: https://doi.org/10.1080/07352689.2022.2093085
[45] Cachi, A. M. (2011). Incompatibilidad polen-pistilo en cerezo (prunus avium l). Caracterización de nuevas fuentes de autocompatibilidad [Tesis, Universidad de Zaragoza]. Dialnet. https://dialnet.unirioja.es/servlet/tesis?codigo=204897
[46] Halász, J., Pedryc, A. y Hegedűs, A. (2007). Origin and dissemination of the pollen‐part mutated SC haplotype which confers self‐compatibility in apricot (Prunus armeniaca). New Phytologist, 176(4), 792-803. doi: https://doi.org/10.1111/j.1469-8137.2007.02220.x
[47] Kakui, H., Kato, M., Ushijima, K., Kitaguchi, M., Kato, S. y Sassa, H. (2011). Sequence divergence and loss‐of‐function phenotypes of S locus F‐box brothers genes are consistent with non‐self recognition by multiple pollen determinants in self‐incompatibility of Japanese pear (Pyrus pyrifolia). The Plant Journal, 68(6), 1028-1038. doi: https://doi.org/10.1111/j.1365-313X.2011.04752.x
[48] Gu, C., Wang, L., Korban, S. S. y Han, Y. (2015). Identification and characterization of S-RNase genes and S-genotypes in Prunus and Malus species. Canadian Journal of Plant Science, 95(2), 213-225. doi: https://doi.org/10.4141/cjps-2014-254
[49] Dongariyal, A., Dimri, D. C., Kumar, P., Choudhary, A., Jat, P. K., Basile, B., Mataffo, A. et al. (2022). Pollen-Pistil Interaction in Response to Pollination Variants in Subtropical Japanese Plum (Prunus salicina Lindl.) Varieties. Plants, 11(22), 3081.doi: http://dx.doi.org/10.3390/plants11223081
[50] Orlando Marchesano, B. M., Chiozzotto, R., Baccichet, I., Bassi, D. y Cirilli, M. (2022). Development of an HRMA-Based Marker Assisted Selection (MAS) Approach for Cost-Effective Genotyping of S and M Loci Controlling Self-Compatibility in Apricot (Prunus armeniaca L.). Genes, 13(3), 548. doi: http://dx.doi.org/10.3390/genes13030548
Notas de autor
Daniela Mejía y Verónica Baquero se encargaron de la adquisición, análisis e interpretación de los datos, incluyendo el trabajo de campo y laboratorio.
Daniela Mejía y Verónica Baquero redactaron el manuscrito inicial.
Maria de Lourdes Torres, Daniela Mejía y Verónica Baquero hicieron ediciones y redacción final del manuscrito.
Daniela Mejía y Verónica Baquero redactaron el manuscrito inicial.
Maria de Lourdes Torres, Daniela Mejía y Verónica Baquero hicieron ediciones y redacción final del manuscrito.
María de Lourdes Torres realizó la revisión crítica del manuscrito.
Maria de Lourdes Torres, Daniela Mejía y Verónica Baquero hicieron ediciones y redacción final del manuscrito.
bioteclab@usfq.edu.ec
Declaración de intereses

