



Sección A: Ciencias Exactas y Físicas
Nanoestructuras inusuales de ácidos nucleicos basados en ADN G-cuádruple
Unusual Nucleic acid nanostructures based on G-quadruplex DNA
ACI Avances en Ciencias e Ingenierías
Universidad San Francisco de Quito, Ecuador
ISSN: 1390-5384
ISSN-e: 2528-7788
Periodicidad: Bianual
vol. 7, núm. 2, 2014
Recepción: 10 Junio 2015
Aprobación: 03 Octubre 2015
Publicación: 30 Diciembre 2015
Autor de correspondencia: mmendez@usfq.edu.ec

Resumen: Se reporta la preparación de un nano-ensamble basado en estructuras de ADN no canónico. Para la nanofabricación de una estructura basada en ADN el auto ensamblaje de ADN G-cuádruple (aparea- miento de bases tipo hoogsteen) y ADN de doble cadena (apareamiento de bases tipo Watson-Crick) fueron utilizados. En general, un número importante de nanoestructuras se han construido explotando la capacidad de apareamiento de bases del ADN canónico de doble cadena. Hay formas alternativas para construcción utilizando otros elementos de ADN tales como G-cuadruple, motivos I, o ADN de triple cadena. Como prueba de prinicipio, previamente hemos reportado el uso de ADN de doble cade- na (oligonucleótidos de ADN sintéticos cortos) con secciones de sitios no apareados capaz de mediar la formación de secciones tetramoleculares (pruebas G-cuádruple) con la finalidad de ensamblar los componentes en estructuras de alto peso molecular. Gel electroforesis como también microscopia de fuerza atómica muestran la formación de nanofibras. La electroforesis de Gel como el dicroismo circu- lar dan evidencia de la presencia de secciones G-cuádruple. De las imágenes de microscopía de fuerza atómica se estimó que el largo de las estructuras va de 250 a 2000 nm con altitud de 0.45 a 4.0 nm. Aquí presentamos otro ejemplo de tales nanofibras. Sugerimos que metodologías similares pueden ser usa-das para construir nanoestructuras más complejas que saquen provecho de las propiedades de distintos nano-rarezas de ADN para aplicaciones capaces de realizar tareas útiles.
Palabras clave: Ensambles inusuales de ácidos nucleicos, nanofibras, G-cuádruple, nanoestructuras inusuales.
Abstract: A nano-assembly based on non-canonical DNA structures is reported. For the nanofabrication of a DNA-based structure the self-assembly of guanine quadruplex (Hoogsteen base pairing) and double- stranded DNA (Watson-Crick base pairing) was exploited. In general, an important number of nanos- tructures have being build exploiting the base pairing capability of canonical double stranded DNA. There are alternative approaches for construction using other DNA elements such as G-quadruplex, I - motifs, or triplexes. As a proof of principle, we have previously reported the use of duplex DNA (short synthetic DNA oligonucleotides) with sections of miss paired sites able to mediate formation of tetramolecular sections (G-quadruplex DNA prove) in order to ensemble the components into high molecular weight structures. Gel electrophoresis as well as atomic force microscopy show the forma- tion of nanofibers. Gel electrophoresis as well as circular dichroism gave evidence of the presence of G-quadruplex sections. From AFM we estimated that the structures lengths expand from 250 to 2,000 nm with heights from 0.45 to 4.0 nm. Here we present another example of such nanofibers. We sug- gest that similar methodologies can be used to build more complex nano-structures that will exploit the properties of different DNA nano-oddities into functional applications.
Keywords: Unusual nucleic acid assembly, nanofibers, G-quadruplexes, nano-oddity.
Introducción
La Nanotecnología está rápidamente cambiando el paradigma de los sistemas de fabricación de una estrategia “top - down” a una estrategia “bottom-up” [1]. Estos procesos que siguen la estrategia “bottom-up” explotan el “autoensamblaje” del material involucrado. Uno de tales materiales con la habilidad de auto ensamblar- se es el ADN [2, 3]. La creación de sistemas moleculares/nanosistemas capaces de realizar funciones complejas integrando múltiples componentes como es en el caso de toda nuestra tecnología basada en la estrategia tradicional top-down es aún un desafío tecnológico muy importante [1, 4]. Las principales limitaciones son la capacidad de diseñar sistemas que sin una inversión importante de energía o de manipulación excesiva puedan ensamblarse por sí mismos en un sistema capaz de realizar una función compleja. Por el momento aún estamos principalmente en la etapa de encontrar materiales que se auto ensamblan en estructuras dentro de la escala nanométrica que por sí mismos posean ya una aplica- ción tecnológica directa. Aquí presentamos un sistema multicomponente del mismo material (ADN) en lo que podría constituir los bloques de construcción de sistemas nanoscópicos funcionales basados en ADN en la frontera entre un sistema molecular y un nanosistema multi componente. Nuestro bloque de construcción básico consiste en ADN con un elemento de cadena doble con un segmento interno de varias guaninas contiguas en ambas cadenas y un segundo elemento constituido por extremos de cadena simple. Estos nucleótidos (en doble cadena) fueron incubados en solución amortiguadora con potasio y el biomaterial formó especies de alto peso molecular (nanofibras) que contienen segmentos de ADN G-cuádruple sin necesidad de ninguna otra manipulación adicional al control de la temperatura del medio usando un termociclador básico.
El ADN G-cuádruple es una molécula de ADN donde participan guaninas (G), que pueden estar en la misma cadena o en distintas cadenas, para formar una estructura estabilizada por enlaces de hidrógeno con cuatro Gs en un mismo plano, una tétrada (Ver Figura 1) [5]. Una molécula de ADN G-cuádruple tiene al menos dos de estos planos o tétradas apilados consecutivamente uno sobre el otro. La estructura además de ser estabiliza- da por los puentes de hidrógeno es estabilizada por cationes como sodio, o potasio que se encuentran alinea- dos con el espacio en el centro de las cuatro guaninas (una especie de canal rodeado de cuatro átomos de oxígeno, uno por cada guanina) y aproximadamente centra- do con respecto al eje longitudinal de la molécula entre dos tétradas consecutivas de ADN G-cuádruple [6]. En la literatura se ha reportado la fabricación de varias nanoestructuras basadas en ADN G-cuádruple [7, 8]. Uno de los posibles mecanismos de construcción es el auto apilamiento de unidades de G-cuádruple para formar estructuras aproximadamente cilíndricas y helicoidales. Se ha observado que oligonucléotidos (especialmente a altas concentraciones) con varias guaninas consecutivas en su secuencia forman superestructuras donde múltiples bloques de G-cuádruple se auto apilan, ya sea de forma que un bloque se apila sobre el otro, o donde una de las cadenas del G-cuádruple se desalinea o desplaza un “peldaño” dejando una o más guaninas expuestas, permitiéndole ahora a esta subunidad interactuar con la subsiguiente unidad de G-cuádruple [9–11]. Reciente- mente, también se han caracterizados sistemas sencillos en los que se observa una región G-quadruplex y una región de ADN de doble cadena [12]. Sin embargo en todos estos sistemas el tipo de secuencias de ADN involucradas en el ensamblaje es limitado (en cuanto a posibilidad de combinación y uso de múltiples secuencias y complejidad de las secuencias). En el sistema propuesto en este artículo demostramos una metodología que no se restringe a una única secuencia de ADN pero que puede utilizar cualquier secuencia de ADN en tanto contenga una pequeña sección de bases guaninas consecutivas no apareadas en alguna región interna de la molécula. La exploración de sistemas cuyo ensamblaje se pueda guiar por un delicado control de la temperatura permitirá generalizar y ampliar el uso del ADN como material base para nanoestructuras parte de biosensores, nano máquinas y otras aplicaciones [2, 6].
| Nombre | Largo | Secuencia 5’ a 3’ |
| C1A | 39 | ACAGTAGAGATGCTGCTGATTCGTTCATGTGCTTCAAGC |
| C1B | TGTCATCTCTACGACGACTAAGCAAGTACACGAAGTTCG | |
| SQ1A | 39 | CAGTAGAGATGCTGCTGAGGGGGGGGTGTGCTTCAAGCG |
| SQ1B | CTCTACGACGACTGGGGGGGGACACGAAGTTCGCTACTG | |
| C2 | 29 | TCTACGACGACTGGGGGGGGACACGAAGT |
Materiales y métodos
Todos los oligonucleótidos de ADN se adquirieron de Midlan Oligos (Midland, TX, USA) y fueron re suspendidos en agua purificada con un contenido de orgánicos totales menor a 34 .g/L y resistividad de 18.2 MΩ·cm. Se utilizó cloruro de tetrametil amonio (ClTMA), per- sulfato de amonio, mercaptoetanol, MgCl., KCl, tris (hi- drometil) aminometano (TRIS), ácido bórico de grado bioquímico o equivalente. Los experimentos de gel electroforesis y microscopía de fuerza atómica se realizaron como fue previamente reportado por nosotros [13]. Las soluciones amortiguadoras que se utilizaron se reportan en la Tabla 1.
Manejo del ADN
Las secuencias de ADN utilizado se muestran en la Fig 2. Este ADN es sintético y viene en forma liofilizada por tanto fue necesario re suspenderlo en agua desionizada. Debido a la alta dependencia de la estructura de los G- cuádruple en la concentración e identidad de los cationes presentes se utilizó durante todo el trabajo agua o amortiguadores preparados en agua con un contenido de orgánicos totales menor a 34 .g/L y resistividad de 18.2 MΩ·cm como se indicó anteriormente. El procedimiento para realizar precipitación con etanol de ADN es ligeramente distinto al usual utilizado en biología mole- cular porque para la preparación del ADN de doble cadena precursor (“DNA sinapsable” [14, 15]) se necesita utilizar una sal que no promueve la formación de DNA G-cuádruple [6]. Para esto se utilizó una sal de aminas cuaternarias (2.5 M ClTMA) [9]. Para los tratamientos térmicos se utilizó un termociclador básico que permite programar la velocidad de aumento/disminución de la temperatura. El DNA se diluyó en el amortiguador apropiado para la aplicación a realizar (Tabla 1). En general se utilizó un ADN de doble cadena con una sección de bases no apareadas constituidas por Guaninas con la finalidad de estimular, en la presencia de iones de potasio, la formación de una estructura tetramolecular (DNA G-cuádruple). Un modelo aproximado del ADN precursor antes de ser sometido a la presencia de un amortiguador con potasio se muestra en la Figura 2. La lista completa de secuencias utilizadas incluyendo las secuencias control se encuentra en la Tabla 2.
Gel electroforesis no desnaturalizante
La electroforésis se realizó de acuerdo a procedimientos estándar [13]. En el caso de las muestras a analizar con ADN de doble cadena (Ej. SQ1A con su secuencia complementaria SQ1B) se incubaron en presencia de potasio. El potasio se conoce es un excelente estabilizador de secuencias G-cuádruple. En caso de formarse esta clase de estructuras la movilidad de las bandas se reducirá considerablemente cuando se compare con el ADN doble cadena original o con la movilidad de oligonucleótidos de cadena simple incapaces de formar secuencias G-cuádruple como la secuencia control C1A y C1B.
Microscopía de Fuerza Atómica
El instrumento utilizado para obtener las imágenes fue un Veeco AFM Nanoscope V. Todas las imágenes fueron adquiridas en “Tapping mode”. A partir de las mues- tras utilizadas para la electroforésis de Gel nativa se prepararon las muestras para los analisis de AFM. Debido a que se necesitan altas concentraciones para el gel electroforesis se necesitó diluir con agua desionizada las muestras hasta concentraciones apropiadas para AFM (0.16 ng/.L). Como substrato se utilizó un wafer de silicio (Silicon Valley Microelectronics, Inc. (Santa Clara, CA, USA).
Construcción del modelo 3D de ADN
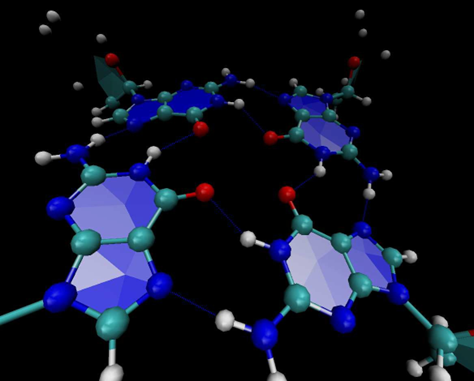
Se utilizó como punto de partida una estructura tipo cierre en base a la estructura depositada en el Protein Data
Bank de código 376D [16]. Para la creación de la región de doble cadena se utilizó la herramienta en línea 3D-DART [17]. El ensamble de la estructura se usó utilizando herramientas del software VMD para la preparación del sistema, incluyendo adición de solvente, de contraiones, y condiciones de borde. Las figuras mostradas corresponden a los resultados de un proceso de optimización de geometría en presencia de solvente y iones.
Resultados y Discusión
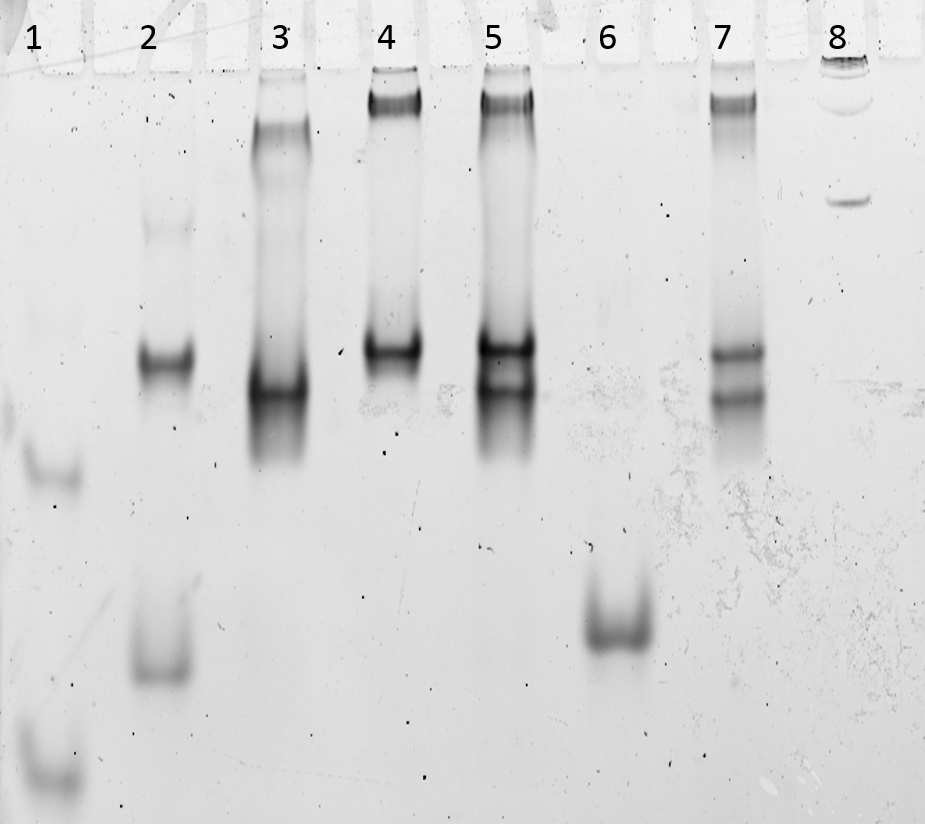
En resumen, se logró ensamblar nanofibras de ADN a partir de elementos de ADN que consistian de una hebra de doble cadena, con una sección desapareada rica en Guaninas en el centro de la estructura y dos terminales de cadena simple a cada lado de la estructura. Las distintas pruebas sugieren que el mecanismo de ensamblaje incluye intermediarios de estructura G.cuádruple. Para llegar a estas nanofibras, se siguió una secuencia cuidadosa de caracterización de cada elemento. Como primer control para explorar la formación de especies G-cuádruple se utilizó la secuencia C2 (ver Tabla 2) que se diseñó para ser complementaria a nuestra secuencia de prueba SQ1A (una de las dos secuencias que constituirá al final las nanofibras). C2 es un olignucleotido que tiene 10 bases menos que la secuencia de prueba SQ1A. Por tanto, la formación de especies G-cuádruple a partir de solo secuencias C2 y solo secuencias SQ1A se podrán fácilmente distinguir entre sí y de las formadas a partir de un intermediario de doble cadena SQ1A con C2. Por ejemplo, la secuencia (C2). tendrá una movilidad en el gel distinta que la especie (C2).SQ1A. Esto nos permitió distinguir si las especies obtenidas se forman a través de lo que denominaremos la vía del ADN- sinapsable como intermediario versus la vía en la que simplemente las cadenas se juntan al azar en la solución. La vía a través del ADN sinapsable es la ruta en que primero se forma un DNA de doble cadena entre dos cadenas complementarias (Ej SQ1A con SQ1B) y a continuación dos moléculas de ADN de doble cadena interactúan entre sí para formar una nueva especie tetramolecular. El nombre “DNA-sinapsable” se originó debido a que esta vía nos recuerda el proceso en biología denominado sinápsis durante la primera fase de la meiosis donde tenemos cuatro hebras de ADN interactuando [14, 15]. Si este proceso de formación vía intermediario de “ADN sinapsable” es el que se siguió durante la formación de nuestras nanofibras, no se pueden formar las especies tipo (C2).SQ1A como intermediarios pues esta combinación indicaría una unión al azar de las cadenas de ADN en la solución. Como se puede ver en la Figura 3 efectivamente no se observan bandas con movilidades que correspondan a especies tipo (C2).SQ1A. En la línea 2 se observan claramente dos bandas de mayor movilidad, la más rápida corresponde- ría a la cadena simple y la siguiente muy posiblemente a una especie G-cuádruple del tipo (SQ1B).. Esta línea 2 nos puede servir de guía para interpretar las otras ban- das. Adicionalmente vale señalar la presencia de bandas de movilidad menor en varias de las líneas del gel e incluso un poco de retención de ADN en los posillos lo que debería corresponder a las nanofibras o sus precursores oligoméricos de mayor tamaño/peso molecular que las cadenas simples de ADN o los correspondientes G-cuádruples. Finalmente es necesario señalar que las secciones de cadena simple en los extremos de los ADN precursores de doble cadena no son complementarios con ninguna secuencia de cadena simple libre en el ADN precursor. Sin embargo, se diseñó la secuencia de esta forma con la finalidad de en un futuro utilizar estos segmentos como puntos de anclaje o de unión a otros tipos de nanoestructuras en base a ADN.
El ADN fue teñido utilizando SByr Green I. Las mues- tras en el gel (Figura 3) en orden de izquierda a derecha incluyen línea 1. 2.0 .M C2, línea 2. 2.0 .M SQ1B, lí-
nea 3. 1.0 .M duplex C2 + SQ1A, línea 4. 1.0 .M de ADN de doble cadena SQ1, línea 5. 1.0 .M C2 + SQ1A y 1.0 .M SQ1, línea 6. 2.0 .M C1 A. línea 7. 2.40 .M C2 + SQ1A a y 2.4 .M SQ1. Línea 8. 100 bp DNA
ladder. El gel se corrió en 0.10 KMgTb, pH 7.8.
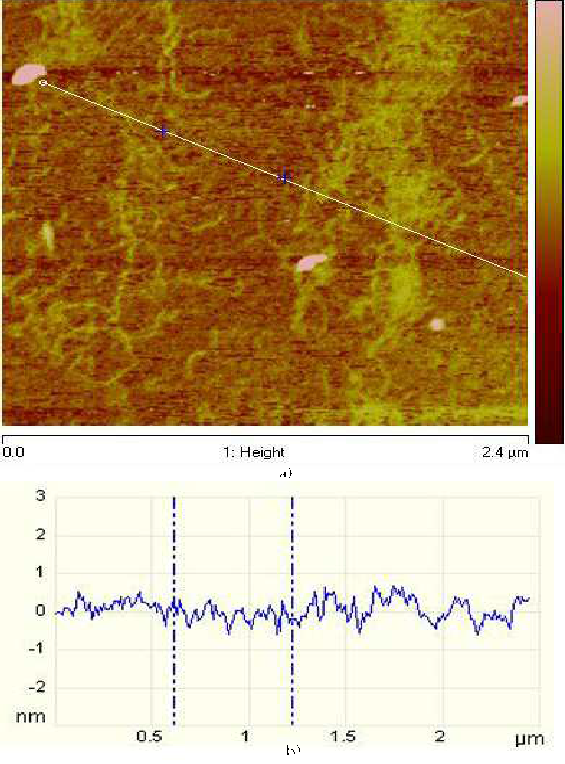
Una vez que se caracterizó los precursores de las nanofibras y dada la presencia de especies de alto peso molecular, posiblemente fibras o sus precursores, se procedió a adquirir imágenes de AFM para identificar la presencia de estas nanoestructuras, así como caracterizar su morfología. Como se observa en la Figura 4, una muestra en base a SQ1A y SQ1B a una concentración de 0.0067 .M por cuádruple cadena, calentada a 90 ◦C por 5 minutos y mantenida a 50 ◦C por 72 h muestra la presencia de especies fibrilares. Bajo otras condiciones también hemos observado la formación de nanofibras [13]. Considerando que las unidades fundamentales solo tienen 39 nucleótidos, el tamaño de estas fibras indica el ensamble de varias decenas de unidades como constituyentes de estas nanofibras. No se escapa a nuestro diseño del esquema original que este material deja puertas abiertas y accesos para combinar las nanofibras con otros segmentos nanométricos en base a ADN u otros materiales como nanopartículas metálicas.
A futuro, se puede conseguir un control de la morfología final de las fibras a través de un control de la temperatura, identidad y concentración de iones en las soluciones amortiguadoras a usar potencialmente pueden controlar la longitud y número de fibras a formarse. Comparando los resultados que hemos reportado previamente [13], y las condiciones aquí descritas observamos que efectiva- mente un cambio en condiciones experimentales afecta profundamente la morfología (tamaño, distribución, longitud) de las nanofibras. Finalmente, considerando la potencial importancia de estructuras de autoensamblaje de ADN en nanotecnología [2, 18], los resultados reportados en el presente trabajo muestran una vía sencilla para combinar dos tipos muy distintos de estructuras en base a ADN (con sus propias características y propiedades como biomateriales) en una misma nanoestructura. El método tiene potencial impacto para la nano fabricación de andamiaje para la creación de nanoestructuras funcionales más complejas.
Agradecimientos
El autor agradece a Veronika Szalai en cuyo laboratorio (en UMBC) se realizaron los experimentos y a la facilidad de microscopía Keith R. Porter de University of Maryland, Baltimore County donde se analizaron las muestras por microscopía de fuerza atómica.
También se agradece a Dennisse Vallejo por la construcción del posible modelo 3D correspondiente a un DNA zipper con las secuencias SQ1A/SQ1B.
Referencias
[1] Ariga, K. 2015. “Nanoarchitecntonics: a new materials horizon for nanotechnology”. Materials Horizon, 2(10): 406-413.
[2] Chen, Y.; Groves, B.; Muscat, R.A.; Seeling, G. 2015. “DNA nanotechnology from test tube to cell”. Nature Nanotechnology, 10(9): 748-760.
[3] Yang, D.; Hartman, M. R.; Derrien, T. L.; An, D.; Yan- cey, K. G.; Cheng, R.; Ma, M.; Luo, D. 2014. “DNA Ma- terials: Bridging Nanotechnology and Biotechnology”. Acc.Chem. Res., 47(6): 1902-1911.
[4] Ouldridge, T. E. 2015. “DNA nanotechnology: unders- tanding and optimisation through simulation”. Molecu- lar Physics, 113(1): 1-15.
[5] Neidel, S. 2009. “The structures of quadruplex mucleic acids and their drug complexes”. Curr. Opin. Struct Biol., 19(3): 239-250.
[6] Bates, P.; Mergny, J. L.; Yang D. 2007. “Quartets in G- major. The First International Meeting on Quadruplex DNA”. EMBO Rep, England, 8: 1003-10.
[7] Dutta, K.; Fujimoto, T.; Inoue, M.; Miyoshi, D.; Sugi- moto, N. 2010. “Development of new functional nanos- tructures consisting of both DNA duplex and quadru- plex”. Chem. Commun., 46(41): 7772-7774.
[8] Nakatsuka, K.; Shigeto, H.; Kuroda, A.; Funabashi, H. 2015 “A split G-quadruplex-based DNA nano-tweezers structure as a signal-transducing molecule for the ho- mogeneous detection of specific nucleic acids”. Biosens Biolectron., 15(74): 222-226.
[9] Mendez, M. A.; Szalai, V. A. 2009. “Fluorescence of unmodified oligonucleotides: A tool to probe G- quadruplex DNA structure”. Biopolymers, 91(10): 841- 850.
[10] Miyoshi, D.; Karimata, H.; Wang, Z. M.; Koumoto K.; and Sugimoto N. 2007. “Artificial G-Wire Switches with 2,2’-Bipyridine Units Responsive to Divalent Me- tal Ions”. J. Am. Chem. Soc., 129: 919-5925.
[11] Alberti, P.; Bourdoncle, A.; Sacca, B.; Lacroix, L. and Mergny, J. L. 2006. “DNA nanomachines and nanostructures involving quadruplexes”. Org. Biomol. Chem., 4: 3383-3391.
[12] Fottichia, I.; Amato, J.; Pagano, B.; Novellino, E.; Petraccone, L.; Giancola, C. 2015. “How are thermodynamically stable G-quadruplex-duplex hybrids?”. J. Therm. Anal. Calorim., 121: 1121-1127.
[13] Mendez, M. A and Szalai, V. A. 2013. “Synapsable quadruplex mediated fibers”. Nanoscale Research Letters, 8: 210.
[14] Fahlman, R. P.; Dipankar, Sen. 1999. “Synapsable DNA Double Helices: Self-Selective Modules for Assembling DNA Superstructures”. J. Am. Chem. Soc., 121(48): 11079-11085.
[15] Fahlman, R. P.; Sen, D. 2010. “Cation-regulated self- association of “synapsable” DNA duplexes”. J. Mol. Biol., 280(2): 237-244.
[16] Shepard, W.; Cruse, W. B.; Fourme, R.; de la Fortelle, E. and Prange, T. 1998. “A zipper-like duplex in DNA: the crystal structure of d(GCGAAAGCT) at 2.1 A resolution”. Structure, 6: 849-861.
[17] van Dijk, M.; Bonvin, A. 2009. “3D-DART: a DNA structure modelling server”. Nucl. Acids Res., 37: W235-W239.
[18] Sadhasivam, S. and Yun, K. 2010. “DNA self-assembly: prospectus and its future application”. Journal of Mate- rials Science, 45(10): 2543-2552.
Notas de autor
mmendez@usfq.edu.ec

