Caracterización del microbioma de plantas de banano (Musa × paradisiaca L.) bajo sistemas de producción orgánico y convencional
Characterization of the banana (Musa × paradisiaca L.) microbiome under organic and conventional production systems
ACI Avances en Ciencias e Ingenierías
Universidad San Francisco de Quito, Ecuador
Recepción: 09 Junio 2021
Aprobación: 01 Junio 2022
Resumen: El cultivo de banano (Musa × paradisiaca L.) es una de las actividades agrícolas más importantes para muchos países y es la principal fruta consumida mundialmente. Lafertilización química es una de las prácticas más comunes utilizada para aumentarel rendimiento y productividad de los cultivos. Una gran cantidad de literaturaha examinado los cambios en las propiedades del suelo asociados con diferentesregímenes de fertilización. A pesar del papel fundamental del microbioma en la saludy la productividad de las plantas, los efectos de los diferentes sistemas de gestiónagrícola sobre las comunidades microbianas son poco estudiados. Este estudioinforma sobre la estructura, diversidad y riqueza de la comunidad microbiana del suelo,rizosfera y hoja de las plantas de banano bajo manejo orgánico y convencional. Seobtuvieron muestras de dos plantaciones ubicadas en El Oro, una de las provinciascon mayor productividad bananera en Ecuador. El análisis se basó en la secuenciaciónde la región V3-V4 del gen del ARNr 16S para bacterias y la región ITS2 para hongos,para luego generar ASVs, que son secuencias de ADN diferenciadas entre sí porun único cambio de nucleótidos, lo que nos permitió caracterizar y distinguir a losmicroorganismos. Se encontró que el sistema de manejo agrícola tuvo un efectosignificativo en la composición y diversidad de la población bacteriana y fúngica. Entérminos generales, la riqueza y uniformidad de la comunidad microbiana fue mayoren el suelo y rizosfera bajo manejo convencional en comparación con el orgánico. Elsuelo y la rizosfera bajo agricultura orgánica se asociaron con una mayor abundanciade Proteobacteria, Firmicutes y Bacteroidetes, y exhibieron una sobrerrepresentaciónde géneros microbianos conocidos como promotores del crecimiento de plantas, asícomo géneros involucrados en procesos importantes del ecosistema como el ciclo de carbono. También encontramos que varios microorganismos del mismo género responden diferente a los dos tipos de manejo agrícola en el suelo y la rizosfera. Por otrolado, las comunidades bacterianas en las hojas fueron más similares en ambos tipos demanejo. Comprender cómo los sistemas de gestión de cultivos a largo plazo modificanla diversidad y la estructura microbiana, como se presenta en esta investigación, puedeayudar a diseñar sistemas agrícolas que puedan mantener una alta rentabilidad de loscultivos de banano mediante la estimulación de bacterias promotoras del crecimientoy supresoras de las enfermedades transmitidas por el suelo.
Palabras clave: diversidad, microorganismos del suelo, agroecosistema, manejo agrícola, banano, microbioma vegetal.
Abstract: Banana (Musa × paradisiaca L.) is one of the most important farming crops in manycountries around the world, while also being the most consumed fruit worldwide. Inorder to increase the yield and productivity of the crops, chemical fertilization is widelyused as a common practice. A variety of literature has examined how soil propertieschange with different methods of fertilization. While the microbiome of plants has animportant role in both health and productivity, few studies focus on the effects of usingdifferent agricultural management on microbiome communities. This study reports onthe structure, diversity, and richness of the microbiome community in soil, rhizosphere,and leaves from banana plants under organic and conventional management. Sampleswere obtained from two plantation sites located in El Oro, a province in Ecuadorwere banana production is among the highest in the country. Analyses were basedon sequencing the region V3-V4 from 16s rRNA gene for bacteria, and ITS2 region forfungi, in order to generate ASVs, DNA sequences with only one nucleotide changethat differentiates them between each other, thus permitting characterization anddistinction of microorganisms. The study found that the agricultural managementshad a significative effect in the composition and diversity of the bacterial and fungipopulation. In general terms, the richness and uniformity of the microbial communitywas higher in the soil and rhizosphere under conventional management in comparisonto the organic. The soil and rhizosphere using the organic management showedassociation with higher abundance of Proteobacteria, Firmicutes and Bacteroidetes, andexhibited an overrepresentation of microbial genera known as plant growth promotersand genera involved in important carbon cycle processes for the ecosystem. It wasalso found that various microorganisms from the same genera had different responsesto both agricultural managements in soil and rhizosphere. On the other hand, thebacterial communities from leaves were more similar in both types of agriculturalsystems. Comprehension on how the methods of long-term cropping managementsystems modified the microbial diversity and structure, as shown in this study, can help design methods in agriculture with high profitability from banana cultivation bystimulating growth promoter bacteria and soil-borne disease suppressors.
Keywords: diversity, ecosystem functions, soil micro-organism, agroecosystem, agricultural management, banana, plant microbiome.
INTRODUCCIÓN
Entre 1969 y 2015 hubo una expansión del uso de la tierra y recursos naturales destinados a la agricultura. Esto fue impulsado por las tecnologías, industrialización y globalización de la agricultura en la Revolución Verde cuando la productividad de los cultivos mejoró significativamente, creciendo más del triple [1,2]. Sin embargo, a pesar de dichas mejoras, el rendimiento se ha empezado a ralentizar, pues existe una gran presión sobre los suelos y recursos hídricos que impiden que la frontera agrícola pueda expandirse aún más [3]. Esto supone una amenaza para el abastecimiento de alimentos en cantidades ideales para una población en aumento, pues para el 2050 se estima que la población humana alcanzará los 9700 millones de personas [1].
El cultivo de banano es una de las actividades agrícolas de mayor importancia para la economía y seguridad alimentaria de varios países incluido el Ecuador. En el 2019 la superficie cosechada de banano en Ecuador fue de 183 mil hectáreas y se exportó cerca de 6,6 millones de toneladas. Las provincias de Los Ríos, El Oro y Guayas suman el 84% de la superficie total cosechada de este producto [4,5]. Sin embargo, así como todos los demás cultivos, el banano es susceptible a una serie de plagas y enfermedades que constituyen un factor limitante para su producción ocasionando grandes pérdidas económicas. Para combatir dichas amenazas se ha empleado fungicidas preventivos como mancozeb o clorotalonil y otros sistémicos como benzimidazoles, triazoles, morfolinas y estrobilurinas, que se aplican en las plantaciones con ayuda de avionetas [6]. El aumento del uso de estos químicos junto con el aporte de fertilizantes sintéticos ha incrementado la productividad del cultivo de banano [7], a costa de una creciente preocupación sobre los impactos negativos en el ambiente y salud de los trabajadores. Adicionalmente, este manejo agrícola convencional está relacionado con la contaminación de aguas subterráneas, pérdida de biodiversidad, aumento de emisiones de gases de efecto invenadero y deterioro de la función del suelo [8,9].
Actualmente se busca lograr una transición hacia una agricultura que optimice las técnicas y recursos disponibles, reduciendo los impactos sobre el ambiente, y permita el desarrollo y mayor rendimiento de los cultivos. La agricultura orgánica se ha asociado a una mayor fertilidad del suelo y un alto rendimiento agrícola, con un menor impacto en el ambiente [10]. En Ecuador, en el 2018 el banano orgánico representó el 7,2% de las exportaciones y para el 2020 este aumentó a 10%. En este contexto las exigencias sanitarias de diferentes países y la tendencia saludable que cada día toma más fuerza entre los consumidores han permitido el crecimiento de la demanda del banano orgánico en el mundo [11]. Sin embargo, aún se necesita una visión más amplia sobre todos los componentes de los sistemas de producción.
Las comunidades microbianas están involucradas en una serie de servicios ecosistémicos modulando el ciclo de nutrientes e interacciones simbióticas y patógenas con las plantas, por lo que su rol en la productividad de los agroecosistemas es esencial [12,13]. Varios estudios han demostrado que la intensificación agrícola puede afectar al microbioma asociado a las plantas y como consecuencia a su rol ecosistémico [14,15]. Dada la importancia de los microorganismos en la agricultura, explorar el efecto de las prácticas agrícolas sobre la diversidad y la estructura microbiana sirve para tener un mejor entendimiento de los procesos ecosistémicos, y, de esta forma, desarrollar sistemas de agricultura sostenible. Para esto, las tecnologías de secuenciación masivapresentan grandes oportunidades ya que permiten detectar una gran proporción demicroorganismos cultivables y no cultivables [16]. El secuenciamiento de amplicones,en este caso 16s rDNA e ITS2, permite la obtención de ASVs (por sus siglas en inglés “Amplicon Sequence Variant”), que son secuencias de ADN únicas que se distinguenentre sí por un único cambio de nucleótidos. Estos se utilizan para clasificar grupos deespecies encontrando variaciones biológicas y ambientales para finalmente determinar patrones ecológicos [17].
Con base en investigaciones previas llevadas a cabo en sistemas agrícolas similares, tenemos la hipótesis de que el tipo de manejo agrícola impactará significativamente las estructura, riqueza y uniformidad de comunidades fúngicas y bacterianas asociadas al banano. Para probar esta hipótesis, en este estudio se caracterizó el microbioma asociado a la rizosfera, suelo y hoja de plantas de banano, mediante secuenciación de amplicones, en dos plantaciones en la provincia del Oro: bajo sistema de producción orgánico y convencional. Para nuestro conocimiento este es el primer estudio en banano que evalúa el efecto del sistema agrícola enfocado en tres compartimentos de la planta.
MATERIALES Y MÉTODOS
Selección de sitio y recolección de muestras
El muestreo se llevó a cabo en dos haciendas bananeras localizadas al sur del Ecuador en el cantón Santa Rosa, provincia de El Oro, en mayo 2019. La hacienda “Central, San José” (3,355000, -79,901611) con manejo fitosanitario orgánico desde hace 5 años, se caracteriza por usar controladores biológicos, composta y fertilizantes orgánicos. La hacienda “Cueva” (-3,359361, -79,916194) con manejo convencional por 60 años, utiliza fungicidas, insecticidas y fertilizantes sintéticos, con un incremento en la frecuencia de aplicaciones de dichos químicos (mensual) a lo largo de los años.
La recolección de muestras se realizó en los días de cosecha. Dentro de cada hacienda se seleccionaron plantas ubicadas en el centro de las plantaciones a una distancia 50 m entre sí, con mínimo 5 hojas. Las muestras de suelo se recolectaron de una ubicación al azar alrededor de la planta a una distancia de 50 cm y una profundidad de 20 a 30 cm. Para obtener las muestras de rizosfera, las raíces se retiraron del suelo con una pala a una profundidad de 10 a 15 cm y luego se agitaron suavemente para limpiar la tierra que no estaba firmemente adherida, para su posterior procesamiento. Finalmente, para las muestras de las hojas, se seleccionó el tercio medio de la cuarta hoja. En total se obtuvieron cuatro muestras de suelo, cuatro de rizosfera y cuatro de hojas. Todas las muestras se colocaron en bolsas de polietileno y se guardaron en una nevera portátil hasta su transporte al laboratorio. En este estudio, los diferentes órganos o compartimientos analizados (suelo, rizosfera y hoja) se denominaron como “fracciones” de la planta.
Procesamiento de muestras y extracción de DNA
Se midieron cuatro parámetros químicos (K, Ca2+, Na+, NO3) y conductividad de la fracción suelo. Para esto se tomó 25 gramos de cada muestra de suelo previamente recolectada y se colocó sobre un filtro, se roció agua destilada sobre la muestra y se recuperarondos gotas (0,6 ml) del líquido resultante para luego colocarlas en los sensores de losmedidores de bolsillo LAQUATwin de HORIBA©.
Las hojas se desinfectaron superficialmente sumergiendo pedazos de hoja de 2 x 2 cm [18] en una solución de NaClO 10% y Tween 0,1% por dos minutos. Los residuos del desinfectante se lavaron con dH2O durante dos minutos [19]. Las muestras se congelaron con nitrógeno líquido y se liofilizaron para luego ser colocadas en una solución enzimática (Macerozima 0,1%, Celulasa 1%, 0,7 M de D-Manitol, 5 mM de MES, 9 μM de Cloruro de calcio dihidratado, 65 μM de Fosfato diácido de potasio, pH 5,7) en una relación 5:1. Se incubó en un agitador a 28 oC, 125 rpm, durante 48 horas. Posteriormente, se realizó tres centrifugaciones seriadas a 500 rcf por 5 min, rescatandoel sobrenadante, y otra a 3000 rcf por 20 min. El pellet obtenido, se resuspendió con PBSy una vez colocado en tubos Eppendorf 1,5 ml, se centrifugó una última vez a 13 000rpm por 1 min. Se descartó el sobrenadante y se conservó a -20 oC [20].
Para obtener la rizosfera, las raíces se colocaron en tubos Falcon con PBS, y se agitó en el vortex por 10 min para desprender la rizosfera. Se extrajeron las raíces del tubo y se centrifugó a 500 rcf durante 10 min. Se descartó el sobrenadante y se conservó a -20 oC [21].
La extracción de ADN de la comunidad microbiana de las tres fracciones de la planta (raíz, suelo y rizosfera) se realizó con el kit comercial DNeasy Powersoil® según las instrucciones del fabricante (Qiagen, Hilden, Alemania). La concentración y calidad del ADN extraído se verificó con el flurómetro Qubit 4 (Invitrogen, Thermo Fisher Scientific, USA) y electroforesis en gel de agarosa.
Preparación de las librerías y secuenciamiento
Se amplificó la región V3–V4 del gen 16S rRNA usando los cebadores 338F (5′-ACTCCTACGGGAGGCAGCA-3′) y 806R (5′-GGACTACHVGGGTWTCTAAT-3′). Se añadieron dos barcodes y seis cambios en marcos de lectura en el extremo 5’ de los cebadores [22]. Cada reacción de PCR se realizó por triplicado, cada una incluyó una mezcla única de tres cebadores con cambio de marco de lectura para cada plato. El protocolo de PCR se realizó con 5 μl de Kapa Enhancers, 5 μl de Kapa Buffer A, 1,25 μl de cada primer (5 μM) y 0,375 μl de una mezcla de ácidos nucleicos peptídicos bloqueadores de genes de ARNr, 0,5 μl de Kapa dNTPs, 0,2 μl de Kapa Robust Taq (Kapa Biosystems, Wilmington, MA), 8 μl de dH2O y 5 μl ADN. Los ciclos de temperatura fueron: 95 °C durante 60 segundos, 24 ciclos a 95 °C por 15 segundos, 78 °C durante 10 segundos, 50 °C por 30 segundos, 72 °C por 30 segundos y finalmente, 4 °C hasta su uso [22]. Para lapurificación de amplicones se usaron perlas magnéticas AMPure XP (Beckman Coulter,Brea, CA) y luego se cuantificaron con el fluorómetro Qubit 2.0 (Invitrogen, Carlsbad, CA).
Para la amplificación de la región ITS2 se usaron los cebadores ITS1-F (5′- CTTGGTCATTTAGAGGAAGTAA-3′) e ITS2 (5′-GCTGCGTTCTTCATCGATGC-3′). Las muestras de ADN se diluyeron a concentraciones de 3,5 ng μl-1. Se prepararon reacciones por triplicado compuestas por 10 ng de ADN, 1 U de buffer, albumina de suero bovino al 0,3%, 2 mM de Cloruro de magnesio, 200 μM de dNTPs, 300 nM de cada primer, y 2 U de la polimerasa DFS- Taq DNA (Bioron, Ludwigshafen, Alemania). Los ciclos de temperatura fueron: 2 minutos a 94 °C, 25 ciclos a 94 °C por 30 segundos, 55°C durante 30 segundos, 72 °C por 30 segundos,y 10 minutos a 72 °C para finalizar [22]. Los productos de PCR pasaron a una purificación enzimática con exonucleasas. Se realizó una segunda amplificación por triplicado con 3 μlde los productos de PCR purificados y cebadores con barcodes específicos para las muestrascon ciclos bajo las condiciones previamente mencionadas [22]. Los amplicones fueronpurificados con el kit Agencourt AMPUre XP (Beckman Coulter, Krefeld, Germany).
Finalmente, los amplicones de bacterias y hongos se unieron en proporciones iguales y fueron diluidas hasta obtener una concentración de 10 pM por secuenciamiento, mismo que se llevó a cabo en la plataforma de Illumina MiSeq (Illumina, San Diego, CA) usando el kit 600-cycle V3 [22], en las instalaciones de la Universidad de Carolina del Norte.
Análisis bioinformático
Las secuencias obtenidas de la región 16S se clasificaron por muestras según sus códigos de identificación y se eliminaron adaptadores y cebadores usando MT-Toolbox [23]. Las secuencias procesadas se filtraron en Sickle eliminando aquellas de baja calidad (Q scores < 20) y con lecturas menores a 1000 [24]. Las secuencias resultantes se colapsaron en ASVs con ayuda del paquete DADA2 en R v4.0.2. Dichos ASVs se clasificaron taxonómicamente usando el método naïve Bayes kmer contra la base de datos Silva v132 generando finalmente la tabla de ASVs [25]. Las secuencias de la región ITS2 se procesaron de igual manera. Sin embargo, para la asignación taxonómica de estas secuencias se utilizó la base de datos UNITE con el paquete MOTHUR [25]. Se obtuvo un total de 233 595 lecturas de la región V3-V4 16s rRNA para bacterias y 1670504 lecturas de la región ITS2 para hongos de 12 muestras (dos tipos de manejo, tres fracciones de la planta, dos réplicas). Luego de normalizar los datos, 28 800 lecturasde bacterias y 30 0000 de hongos fueron retenidas para el análisis. Las secuencias de altacalidad generaron 2190 ASVs para bacterias y 1118 ASVs para hongos. Todos los datos seencuentran depositados en https://github.com/darioxr/ACI_banano_cultivo
Análisis estadísticos
Se comparó las comunidades microbianas entre las muestras usando tres métricas amplias: diversidad, composición y abundancia con los paquetes OhChibi y vegan en R. Se calculó Chao1, Inverso de Simpson e Eveness de las comunidades de bacterias y hongos como medida de diversidad alfa. Asimismo, se probó la similitud de la composición microbiana de las múltiples fracciones usando un análisis de coordenadas principales (PcoA) junto con PERMANOVA para calcular la variación en la β-diversidad explicada por la variable fracción. Para maximizar la discriminación entre los diferentes sistemas agrícolas, orgánico y convencional, se usó un análisis canónico de coordenadas principales (CAP) en base a las matrices de disimilitud Bray-Curtis. Se llevaron a cabo ordenaciones separadas para bacterias y hongos. También se analizaron las abundancias diferenciales para explorar que taxones contribuyen a la variación en la comunidad, tanto a nivel de filo para bacterias como a nivel de clase para hongos [26,27]. El paquete VennDiagram se utilizó para construir diagramas de Venn de los dos tipos de manejo por cada fracción para observar el número de ASVs únicos y compartidos entre los diferentes sistemas agrícolas [28]. Finalmente, se realizaron mapas de calor usando el paquete pheatmap, para explorar cambios en las comunidades microbianas en cada fracción según el tipo de manejo a una escala taxonómica más fina [29].
RESULTADOS
Sistemas de manejo agrícola y propiedades físico-químicas del suelo
Se encontró que la concentración de los parámetros K, Na+, NO3 , (ppm) y conductividad (µS/cm) fueron mayores en el sistema agrícola convencional, mientras que la concentración de Ca2+ (ppm) fue ligeramente mayor en el manejo orgánico (Tabla 1).
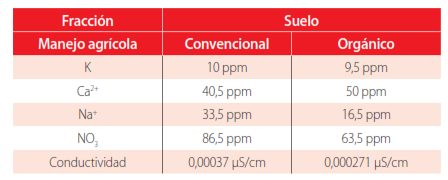
Concentración de los parámetros físico-quimicos de las muestras de suelo analizadas.
Efecto de las diferentes fracciones y regímenes de fertilización sobre la diversidad alfa
Tanto la diversidad alfa de las comunidades de bacterias como de hongos, se vio afectada según el órgano de la planta analizado (fracción). Los índices de diversidad Chao1, Inverso de Simpson e Eveness fueron mayores en la rizosfera, seguida del suelo y finalmente la hoja, a excepción del Inverso de Simpson en la comunidad de los hongos el cual fue mayor en suelo (Tablas 2–3). Por otro lado, el efecto del tipo de manejo agrícola sobre la diversidad alfa de las comunidades bacterianas y fúngicas se analizó por separado para cada fracción. Se observó que los tres índices analizados mostraron consistentemente una mayor diversidad alfa en el manejo convencional en comparación con el orgánico, en las fracciones rizosfera y suelo. Al evaluar las hojas, los índices Inv Simpson e Eveness, excluyendo a Chao1, respaldaron una mayor diversidad bacteriana en el manejo orgánico, mientras que en el caso de la comunidad fúngica de la hoja dichos índices fueron mayores en el manejo convencional (Tablas 2–3).
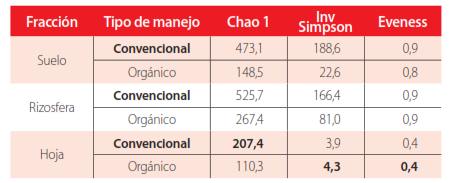
Parámetros de α-diversidad para la comunidad de bacterias determinados para los dos tipos de manejo dentro de cada fracción.
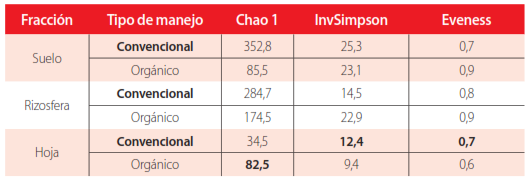
Parámetros de α-diversidad para la comunidad de hongos determinados para los dos tipos de manejo agrícola dentro de cada fracción.
Efecto de las diferentes fracciones y regímenes de fertilización sobre la diversidad beta
Los cambios en la diversidad beta de la comunidad microbiana fueron evaluados calculando las disimilitudes de Bray-Curtis. Se encontró que las comunidades bacterianas se separaron en clusters diferentes según los órganos de la planta. La coordenada 1, que representó el 29,9% de la variación, separó las fracciones subterráneas (rizosfera y suelo) de la aérea (hoja). El análisis PERMANOVA mostró que la variable fracción tuvo un efecto significativo (p<0,05) sobre la composición de la comunidad de bacterias asociadas a las plantas de banano explicando el 43% de la variación (Fig. 1A).
En el caso de la comunidad fúngica, este efecto fue menor pero igualmente significativo (p<0,05) y se observó que los clusters de las distintas fracciones se encuentran más dispersos. El PERMONOVA corroboró las observaciones mostrando que la variación explicada por la variable fracción fue de 26% (Fig. 1B).
Mediante análisis canónico de coordenadas principales (CAP), se observó que el tipo de manejo agrícola tuvo un efecto significativo (p<0,05) sobre las comunidades de bacterias y hongos, y fue mayor para los hongos con 11% de varianza en comparación con el 9,3% de varianza para la comunidad bacteriana. Al observar las gráficas, se identifican dos clusters según el tipo de manejo separados por la coordenada 1 para ambas comunidades microbianas (Fig. 1C–1D).
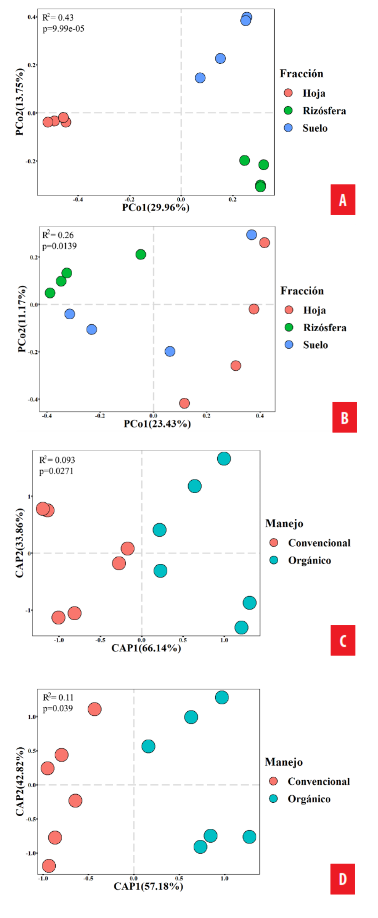
Figura 1.
Efecto del tipo de la fracción y tipo de manejo en la estructura de las comunidades bacterianas yfúngicas. Ordenaciones PCo en base a disimilitudes Bray-Curtis para A) bacterias y B) Hongos. La varianzaexplicada por cada coordenada está colocada en paréntesis. Ordenaciones CAP de comunidades C) de bacteriasy D) de hongos maximizando la discriminación entre los diferentes sistemas agrícolas, orgánico y convencional.La correlación canónica de cada eje CAP se da entre paréntesis. La varianza explicada se encuentra en la esquinasuperior izquierda (con 95% de intervalo de confianza, significancia evaluada con 104 permutaciones).
Composición de bacterias y hongos en las diferentes fracciones analizadas y manejos agrícolas
La abundancia relativa de las bacterias fue investigada a nivel de filo. De los 18 filos detectados, los más abundantes, independientemente del tipo de manejo o fracción, fueron Proteobacterias (49,6%), Firmicutes (11%), Bacteroidetes (10,5%), Acidobacteria (7,8%) y Actinobacteria (6,6%). La composición de filos fue similar para el suelo y rizosfera, pero en la hoja hubo una abundancia relativa predominante de Proteobacteria (78,4%) y Firmicutes (19,3%).
Dentro de cada fracción hubo cambios en la distribución de filos en función de los diferentes tipos de manejo. El manejo orgánico aumentó la abundancia relativa de Proteobacteria en el suelo y rizosfera, pero disminuyó en la hoja en comparación con el convencional. El filo Firmicutes se encontró en mayor proporción en el suelo y hoja orgánica, y en la rizosfera convencional. El manejo orgánico enriqueció a Bacteroidetes en las tres fracciones, mientras que Acidobacteria, Actinobacteria, Patescibacteria y Chloroflexi aumentaron en el suelo y rizosfera convencional (Fig. 2A). Por otro lado, el manejo convencional tuvo más cantidad de variantes de secuencia de amplicones (ASVs) o microorganismos únicos asociados a este manejo en el suelo (723 ASVs) (Fig. 32B), rizosfera (621 ASVs) (Fig. 2C) y hoja (111 ASVs) (Fig. 2D) en comparación con el orgánico (247, 348 y 58 ASVs respectivamente). La fracción con la mayor cantidad de ASVs compartidos entre ambos manejos fue la hoja (24%) seguido de la rizosfera (9,9%) y finalmente el suelo (2,7%). Finalmente, a una escala taxonómica más fina (Fig. 3) se observó que el manejo orgánico en el suelo y rizosfera tiende a enriquecer a la mayoría de ASVs, mientras que en las hojas el enriquecimiento de ASVs fue similar en ambos manejos.
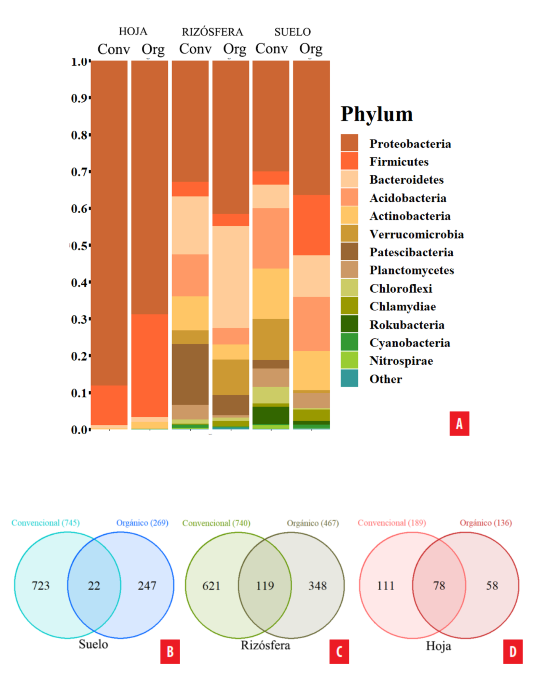
Figura 2.
Composición filogenética bacteriana asociada a los diferentes manejos agrícolas en cada fracción de la planta. Abundancia relativa de A) los filos bacterianos presentes en los manejos agrícolas, convencional (conv) y orgánico (org), en la hoja, rizosfera y suelo. Diagrama de Venn con el número de ASVs compartidos y únicos de cada manejo agrícola, orgánico y convencional, en B) el suelo C) la rizosfera y D) la hoja.

Figura 3.
Heatmap con la asignación taxonómica para cada ASV de los diferentes tipos de manejo asociados a A) elsuelo, B) la rizosfera y C) las hojas. El heatmap muestra los recuentos de los 40 ASVs más abundantes por muestrapara la comunidad de bacterias. Los ASVs están coloreados según su contribución al recuento total (azul: aportaun alto porcentaje de AVS a la muestra; verde claro: contribuye con un bajo porcentaje de ASVs).
Por otro lado, la abundancia relativa de los hongos fue revisada a nivel de clase, siendo las más abundantes, independientemente de la fracción o manejo, Sordariomycetes (34,6%), Dothideomycetes (17,4%), Eurotiomycetes (11,9%), Agaricomycetes (8,6%) y Mortierellomycetes (6,5%). En este caso las abundancias relativas de las clases varían mucha más entre fracciones y entre manejos agrícolas. En general, la abundancia de Sordariomycetes se vio favorecida por el manejo convencional en las tres fracciones. Dothideomycetes estuvo enriquecido en el suelo, rizosfera y hoja bajo manejo orgánico, llegando predominar en esta última fracción. La hoja y suelo orgánico aumentaron la abundancia de Malasseziomycetes. Finalmente, en la rizosfera, hubo un aumento evidente de la clase Agaricomycetes bajo manejo convencional, mientras que, Mortierellocytes fue muy abundante en el manejo orgánico (Fig. 4A).
En los diagramas de Venn se observó que el manejo convencional tiene más cantidad de ASVs tanto en el suelo (496 ASVs) (Fig. 4B) como en la rizosfera (270 ASVs) (Fig. 4C) mientras que la hoja tuvo más ASVs asociados al manejo orgánico (135 ASVs) (Fig. 4D). La fracción con la mayor cantidad se ASVs compartidos entre ambos manejos fue la rizosfera (20,6%) seguido de la hoja (8,76%) y finalmente el suelo (8,4%). En los mapas de calor a una escala taxonómica más fina, se observó que el enriquecimiento de ASVs es más equitativo entre ambos manejos en las tres fracciones (Fig. 5).
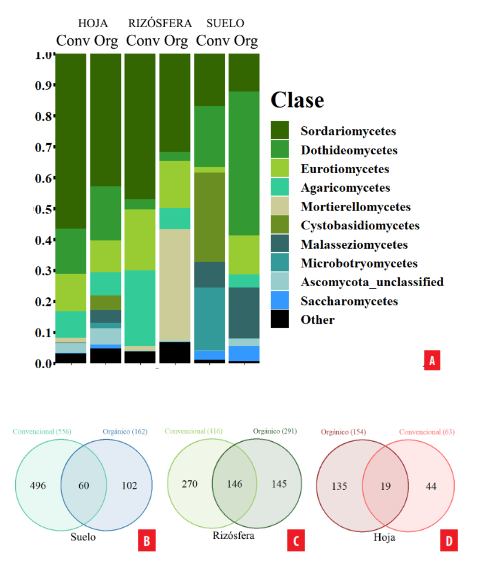
Figura 4.
Composición filogenética fúngica asociada a los diferentes manejos agrícolas en cada fracción de la planta. Abundancia relativa de A) las clases fúngicas presentes en los manejos agrícolas, convencional (conv) y orgánico (org), en la hoja, rizosfera y suelo. Diagrama de Venn con el número de ASVs compartidos y únicos de cada manejo agrícola, orgánico y convencional, en B) el suelo C) la rizosfera y D) la hoja.

Figura 5.
Heatmap con la asignacióntaxonómica para cada ASV de los diferentes tiposde manejo asociados a A) el suelo, B) la rizosferay C) las hojas. El heatmap muestra los recuentosde los 40 ASVs más abundantes por muestrapara la comunidad de hongos. Los ASVs estáncoloreados según su contribución al recuentototal (café: aporta un alto porcentaje de AVS ala muestra; amarillo: contribuye con un bajoporcentaje de ASVs).
DISCUSIÓN
Las plantas han evolucionado en conjunto con microorganismos cuyo rol en los agroecosistemas ha sido reportado extensamente en la literatura [30, 31], lo que ha permitido acumular información sobre la estructura y dinámica del microbioma vegetal. Sin embargo, aún no se conoce del todo bien cómo dichas comunidades responden a los diferentes tipos de sistemas agrícolas.
Los resultados sobre las propiedades químicas del suelo varían entre estudios según los sistemas de fertilización y protección de plantas. La química del suelo ha sido considerada como un determinante estadísticamente significativo de la estructura de la comunidad microbiana del suelo [32,15]. Sin embargo, se ha encontrado que el tipo de manejo agrícola no altera de forma significativa el contenido químico del suelo, o que la diferencia de algunos parámetros químicos entre el manejo orgánico y convencional es mínima o no significativa [33,14,34], como se encontró en este estudio; por lo que la probabilidad de que esta diferencia tenga significancia biológica es mínima [32]. Adicionalmente, en previos estudios se ha encontrado que las comunidades microbianas de sistemas de manejo agrícola con el mismo régimen de protección vegetal, mecánico o químico, comparten menos similitud que los sistemas con un estado nutricional similar, pero diferentes regímenes de protección vegetal, lo que sugiere que el componente fitosanitario tiene una importancia secundaria [32]. Por estos motivos, las enmiendas colocadas en los diferentes sistemas de manejo agrícola probablemente tengan un efecto más directo en la estructura de la comunidad microbiana que indirectamente al alterar el estado químico del suelo [35].
Varios de los factores que dan forma a las comunidades de bacterias y hongos incluyen el compartimento del hospedador, los factores ambientales y genotipo del hospedador [36]. El compartimento o fracción de la planta es probablemente la variable que induce el mayor efecto sobre la estructura de las comunidades microbianas [37,38]. En este estudio se encontró que las muestras se agrupan según la fracción de la planta analizada. Las comunidades microbianas de la parte subterránea, es decir del suelo y rizosfera, se asemejan más en comparación con las hojas (Fig. 1A). Esto no es sorprendente si se considera que los microorganismos presentes en la rizosfera se reclutan y acumulan a partir del suelo [39]. A parte de ensamblar una comunidad diferente de bacterias, las hojas tienen menos diversidad que las otras fracciones analizadas (Tabla 2–3), lo mismo que se ha reportado en un estudio en sorgo [40]. Se ha sugerido que existe una disminución progresiva de la diversidad desde suelo a la rizosfera y luego hacia la parte aérea de la planta como las flores y hojas. Además, varios estudios señalan que la comunidad microbiana en las hojas está influida fuertemente por factores específicos del huésped, mientras que los factores del suelo no afectan significativamente a esta fracción de la planta [36].
Otro de los factores igual de importantes al ensamblar las comunidades microbianas es el tipo de manejo agrícola [41]. En este estudio este efecto fue mayor para las comunidades fúngicas en comparación con las bacterias. El establecimiento de hongos en las raíces de las plantas parece verse más afectado por variaciones estocásticas y responder diferente a los factores ambientales en comparación con las bacterias [36] lo que se corroboró a nivel de clase (Fig. 4) y a escalas taxonómicas más finas (Fig. 5).
En general, se considera que el manejo orgánico alberga mayor diversidad microbiana debido a la aplicación de sustratos ricos en nutrientes como la composta y prácticas biológicas sin la interferencia de compuestos sintéticos que ayudan a generar nichos heterogéneos que pueden ser ocupados por una comunidad microbioma altamente variable, mientras que, el estrés inducido por el uso de pesticidas disminuirá la diversidad para inhibir ciertos grupos de microorganismos [42]. Sin embargo, este efecto difiere entrevarios estudios según las metodologías, contextos y variables analizadas. Seha visto, por ejemplo, que el efecto del tipo de manejo agrícola varía en cada órgano y estadio de crecimiento de la planta [37] en diferentes años de muestreo [32,43] ubicaciones geográficas [44,45], e incluso al evaluar diferentes estrategias de secuenciamiento [46]. Adicionalmente, las composiciones y tipos de fertilizantes sintéticos y orgánicos, así como estrategias de manejo varían entre estudios, pues algunos incluyen arado o tratamientos para la salud del suelo [47]. Esto incluso se aplica a estudios de macrobiota, pues se ha encontrado que los resultados varían entre grupos de organismos y paisajes dado que la estructura y heterogeneidad del paisaje son factores que influyen en la biodiversidad de las diferentes áreas geográficas [48]. Por estos motivos, los efectos del tipo de manejo agrícola sobre el microbioma asociados a los cultivos son complejos y diversos, y resulta difícil obtener conclusiones universales válidas.
En este estudio se encontró que la diversidad fue mayor en el suelo y rizosfera bajo manejo convencional. Se ha sugerido que la aplicación intensiva de composta puede llevar a la acumulación de metales pesados lo que llevará a inhibir ciertos microorganismos [49,50]. Así mismo se tiene la hipótesis de que la disponibilidad de un sustrato rico en nutrientes, como los usados en el manejo orgánico, promueve el crecimiento de organismos copiotróficos [32]. Al facilitar la reproducción intensiva de estos microorganismos, muchos otros se verán desplazados por lo que la diversidad se verá reducida, principalmente en términos de uniformidad, dada la predominancia únicamente de ciertos microorganismos. En el caso de las hojas, los índices de diversidad arrojaron diferentes resultados, por lo que no se puede concluir sobre el efecto del tipo de manejo sobre la diversidad bacteriana y fúngica en esta fracción. Esto en parte se debe a que el microbioma de la hoja está mayormente influido por la química y topología del tejido foliar y el ambiente alrededor, y menos limitado por propiedades del suelo [40], esto se ve reflejado incluso en los mapas de calor (Fig. 5) donde el enriquecimiento de los diferentes ASVs es similar en ambos manejos.
Al analizar la composición microbiana, se encontró que la abundancia de los filos Proteobacteria y Bacteroidetes es mayor en suelo y rizosfera bajo manejo orgánico, mientras que los filos Acidobacteria y Actinobacteria fueron más abundantes en el manejo convencional.
Estos resultados concuerdan parcialmente con lo esperado en base al estilo de vida putativo de estos microorganismos. Los filos Proteobacteria, Bacteroidetes y Actinobacteria son principalmente copiotróficos [51], es decir, crecen rápido en ambientes ricos en nutrientes como se considera al manejo orgánico [32]. Por otro lado, el filo Acidobacteria se lo considera oligotrófico, es decir, crecen lento en ambientes pobres en nutrientes [51] por lo que se espera que se encuentren enriquecidos bajo manejo convencional [32]. Sin embargo, no todos los miembros de un grupo taxonómico tienen las mismas características ecológicas [52] y se espera que las respuestas a los diferentes manejos agrícolas ocurran a niveles taxonómicos bajos [42]. En efecto, en los mapas de calor (Fig. 3, 5) se observa que miembros del mismo grupo taxonómico, como Bacillaceae, responden diferente a ambos manejos agrícolas.
La hipótesis general es que la diversidad microbiana de las plantas es crítica para su integridad, funcionamiento y sostenibilidad de los ecosistemas, y una menor diversidad en el manejo orgánico puede ser desafortunada [49], sin embargo, una mayor diversidad es importante solo si está ligada a un componente de funcionalidad ecológica [53]. En este estudio se encontró que el suelo y la raíz bajo manejo orgánico tuvieron menos diversidad microbiana, pero enriquecen más ASVs asociados a funciones ecológicas importantes como es el caso de Acidobacteria, taxón reconocido como keystone en el suelo [54], asociado con la descomposición de materia orgánica lo que implica su importancia en del ciclo de carbono, además de incluir bacterias promotoras de crecimiento vegetal [55]. Burkholderia también se encuentra enriquecido en el suelo y rizosfera bajo manejo orgánico, este taxón tiene especies promotoras de crecimiento vegetal y son capaces de reducir la disponibilidad de metales pesados [56]. Finalmente, el taxón Rhizobiales, enriquecido en el suelo orgánico, ha sido reconocido por sus especies fijadoras de nitrógeno y facilitadoras de fitohormonas, esto en conjunto favorece el crecimiento de las plantas. También se ha reportado su capacidad de degradar contaminantes orgánicos y resistir a la presencia de metales pesados [57]. Por otro lado, al analizar las comunidades fúngicas (Fig. 5), se observa un enriquecimiento de Fusarium en ambos tipos de manejo, género que alberga especies patógenas para varios cultivos incluido el banano [58]. También se observó la presencia de hongos con funciones ecosistémicas importantes como Penicillium involucrado en la solubilización del fosfato [59], este se encuentra enriquecido en las hojas orgánicos donde podría estar jugando un rol de defensa induciendo el desarrollo de resistencia adquirida como se ha observado en hojas de algodón y melón [60].
Este estudio estuvo limitado por variables espacio temporales, se muestreo en un punto de tiempo único a una escala local, por lo que los resultados no se pueden generalizar a todos los sistemas agrícolas. Las conclusiones deben ser confirmadas por experimentos a largo plazo en condiciones ambientales diversas a escalas geográficas y temporales más grandes.
CONCLUSIONES
El tipo de manejo agrícola fue importante al momento de estructurar las comunidades bacterianas y fúngica. En la hoja este efecto fue menor dado que este tejido de la planta se ve más limitado con la genética del huésped, por lo que estudios con esta temática deben enfocarse en el suelo y la rizosfera que se ven más influidos por el tipo de manejo dado que están en contacto directo con las diferentes enmiendas orgánicas e inorgánicas. El manejo convencional albergó mayor diversidad microbiana en el suelo y la raíz en comparación con el orgánico, sin embargo, este último enriqueció más bacterias y hongos con funciones ecológicas importantes.
AGRADECIMIENTOS
Agradecemos especialmente a los agricultores por permitir el acceso a sus tierras de cultivo y proveer la información necesaria. A la Universidad de Carolina del Norte por sus instalaciones para el secuenciamiento. Los fondos de financiamiento que posibilitaron la ejecución de este estudio provienen del Laboratorio de Agrobiotecnología, USFQ.
Esta investigación se ejecutó bajo la autorización del Ministerio del Ambiente del Ecuador por medio del Contrato Marco de Acceso a Recursos Genéticos MAE-DNB- CM-2018-0085 con el título “Estudio de Diversidad de Microorganismos de Importancia Medica, Veterinaria, Industrial, Ambiental y Agropecuaria del Ecuador”.
CONTRIBUCIÓN DE LOS AUTORES
Claudia Zapata, Antonio León y Dario Ramírez concibieron la investigación y realizaron el diseño de la metodología. Claudia Zapata y Dario Ramirez adquirieron, analizaron e interpretaron los datos. Darío Ramírez realizó el secuenciamiento y su posterior procesamiento. Antonio León adquirió los fondos, proveyó los recursos y realizó la supervisión del estudio. Claudia Zapata redactó el manuscrito y Darío Ramírez y Antonio León realizaron críticamente el contenido y validaron los resultados. Alejandra Paladines estuvo involucrada en el trabajo de campo.
CONFLICTO DE INTERESES
Los autores declaran que no existen conflictos de intereses en el presente trabajo.
REFERENCIAS
[1] FAO. (2017). El futuro de la alimentación y agricultura: tendencias y desafíos (I6881ES/1/02.17) http://www.fao. org/3/i6881s/i6881s.pdf
[2] Khush, G. (1999). Green revolution: preparing for the 21st century. Genome 42(4),646-655. doi: https://doi. org/10.1139/g99-044
[3] Paul, J., Choudhary, A., Suri, V., Sharma, A., Kumar, V. y Shobhna (2014). Bioresource nutrient recycling and its relationship with biofertility indicators of soil health and nutrient dynamics in rice–wheat cropping system. Communications in soil science and plant analysis 45(7),912-924. doi: https://doi.org/10.1080/00103624. 2013.867051
[4] FAO. (2020). FAOSTAT. Recuperado el 11 abril del 2020, desde: http://www.fao.org/faostat/es/#data
[5] INEC. (2020). Encuesta de Superficie y Producción Agropecuaria Continua- ESPAC 2019. https://www.ecuadorencifras. gob.ec/documentos/web-inec/Estadisticas_agropecuarias/espac/espac-2019/Presentacion%20de%20los%20 principales%20resultados%20ESPAC%202019.pdf
[6] Marin, D., Romero, R., Guzmán, M. ySutton,T. (2003). Black Sigatoka: an increasing threatto banana cultivation. Plant disease 87(3),208-222. doi: https://doi.org/10.1094/PDIS.2003.87.3.208
[7] Arvanitoyannis, I. y Mavromatis, A. (2009). Banana cultivars, cultivation practices, and physicochemical properties. Critical Reviews in Food Science and Nutrition 49(2),113-135. doi: https://doi. org/10.1080/10408390701764344
[8] Gruber, N. y Galloway, J. (2008). An Earth-system perspective of the global nitrogen cycle. Nature 451(7176),293- 296. doi: https://doi.org/10.1038/nature06592
[9] Yin, C., Fan, F., Song, A., Li, Z., Yu, W. y Liang, Y. (2014). Different denitrification potential of aquic brown soil in Northeast China under inorganic and organic fertilization accompanied by distinct changes of nirS-and nirK- denitrifying bacterial community. European journal of soil biology 65,47-56. doi: https://doi.org/10.1016/j. ejsobi.2014.09.003
[10] Evenson, R. y Gollin, D. (2003). Assessing the impact of the Green Revolution, 1960 to 2000. Science 300(5620),758- 762. doi: https://doi.org/10.1126/science.1078710
[11] Oportunidades y desafíos del mercado internacional para el banano orgánico (2020, Agosto 12). Agrocalidad. https://www.agrocalidad.gob.ec/oportunidades-y-desafios-del-mercado-internacional-para-el-banano-organico/
[12] Bahram, M., Hildebrand, F., Forslund, S., Anderson, J., Soudzilovskaia, N., Bodegom, P. y Bork, P. (2018). Structure and function of the global topsoil microbiome. Nature 560(7717),233-237. doi: https://doi.org/10.1038/s41586- 018-0386-6
[13] Bardgett, R. y Van Der Putten, W.(2014). Below ground biodiversity and ecosystem functioning. Nature 515(7528), 505- 511. doi: https://doi.org/10.1038/nature13855
[14] Pershina, E., Valkonen, J., Kurki, P., Ivanova, E., Chirak, E., Korvigo, I. y Andronov, E. (2015). Comparative analysis of prokaryotic communities associated with organic and conventional farming systems. PLoS One 10(12). doi: https:// doi.org/10.1371/journal.pone.0145072
[15] Bakker, M., Looft, T., Alt, D., Delate, K. y Cambardella, C. (2018). Bulk soil bacterial community structure and function respond to long-term organic and conventional agricultural management. Canadian journal of microbiology 64(12),901-914. doi: https://doi.org/10.1139/cjm-2018-0134
[16] Clooney, A. G., Fouhy, F., Sleator, R. D., O’Driscoll, A., Stanton, C., Cotter, P. D. y Claesson, M. J. (2016). Comparing apples and oranges: next generation sequencing and its impact on microbiome analysis. PloS one 11(2),e0148028. https://doi.org/10.1371/journal.pone.0148028
[17] Callahan, B. J., McMurdie, P. J. y Holmes, S. P. (2017). Exact sequence variants should replace operational taxonomic units in marker-gene data analysis. The ISME journal 11(12),2639-2643. https://doi.org/10.1038/ismej.2017.119
[18] Gamboa, M. A., Laureano, S. y Bayman, P. (2003). Measuring diversity of endophytic fungi in leaf fragments: does size matter? Mycopathologia 156(1),41-45. doi: https://doi.org/10.1023/A:1021362217723
[19] Lu-Irving, P., Harenčár, J., Sounart, H., Welles, S., Swope, S., Baltrus, D. y Dlugosch, K. (2019). Native and Invading Yellow Starthistle (Centaurea solstitialis) Microbiomes Differ in Composition and Diversity of Bacteria. MSphere 4(2). doi: https://doi.org/10.1128/mSphere.00088-19
[20] Jiao, J., Wang, H., Zeng, Y. y Shen, Y. (2006). Enrichment for microbes living in association with plant tissues. Journal of Applied Microbiology 100(4),830–837. doi: https://doi.org/10.1111/j.1365-2672.2006.02830.x
[21] Schmidt, J., Rodrigues, J., Brisson,V., Kent, A. y Gaudin, A. (2020). Impacts of directed evolution and soil management legacy on the maize rhizobiome. Soil Biology and Biochemistry 145,107794. doi: tps://doi.org/10.1016/j. soilbio.2020.107794
[22] Finkel, O., Salas-González, I., Castrillo, G., Spaepen, S., Law, T., Teixeira, P., Jones, C. y Dangl, J. (2019). The effects of soil phosphorus content on plant microbiota are driven by the plant phosphate starvation response. PLOS Biology 17(11),1–34. doi:https://doi.org/10.1371/journal.pbio.3000534
[23] Yourstone, S., Lundberg, D., Dangl, J. y Jones, C.2014). MT-Toolbox: improved amplicon sequencing using molecule tags. BMC bioinformatics 15(1), 1-7. doi: https://doi.org/10.1186/1471-2105-15-284
[24] Joshi, N. y Sickle, F. (2011). NoTitle. A Sliding-Window, Adaptive, Quality-BasedTrimmingTool for FastQ Files (Version 1.33). https://github.com/najoshi/sickle
[25] Callahan, B., McMurdie, P., Rosen, M., Han, A., Johnson, A. y Holmes, S. (2016). DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods 13(7), 581–583. doi: https://doi.org/10.1038/nmeth.3869
[26] Oksanen, J., Blanchet, F., Kindt, R., Legendre, P., Minchin, P., O’hara, R. y Wagner, H. (2016). Vegan: Community Ecology Package. R Package Version. 2.0-10. CRAN. FEMS microbiology ecology 83(3), 607-621. https://cran.r-project. org/soils
[27] Salas, I. (2019). isaisg/ochibi. Github. https://github.com/isaisg/ohchibi
[28] Chen, H. (2018). Package“VennDiagram”. R Package Versión 1.6.20. CRAN. https://cran.r-project.org/
[29] Kolde, R. (2019). pheatmap: Pretty Heatmaps. R Package Version 1.0.12. CRAN. https://cran.r-project.org/
[30] Dos Santos, L. y Olivares, F. (2021). Plant microbiome structure and benefits for sustainable agriculture. Current Plant Biology 100198. doi: https://doi.org/10.1016/j.cpb.2021.100198
[31] Schlaeppi, K. y Bulgarelli, D. (2015). The plant microbiome at work. Molecular Plant-Microbe Interactions 28(3),212- 217. doi: https://doi.org/10.1094/MPMI-10-14-0334-FI
[32] Hartmann, M., Frey, B., Mayer, J., Mäder, P. y Widmer, F. (2015). Distinct soil microbial diversity under long-term organic and conventional farming. The ISME journal 9(5),1177-1194. doi: https://doi.org/10.1038/ismej.2014.210
[33] Li, R., Khafipour, E., Krause, D., Entz, M, de Kievit, T. y Fernando, W. (2012). Pyrosequencing reveals the influence of organic and conventional farming systems on bacterial communities. PloS one 7(12). https://doi.org/10.1371/ journal.pone.0051897
[34] Mäder, P., Fliessbach, A., Dubois, D., Gunst, L., Fried, P. y Niggli, U. (2002). Soil fertility and biodiversity in organic farming. Science 296(5573),1694-1697. doi: https://doi.org/10.1126/science.1071148
[35] Bünemann, E., Schwenke, G. y Van Zwieten, L. (2006). Impact of agricultural inputs on soil organisms—a review. Soil Research, 44(4),379-406. doi: https://doi.org/10.1071/SR05125
[36] Trivedi, P., Leach, J,Tringe, S., Sa, T. y Singh, B. (2020). Plant–microbiome interactions: From community assembly to plant health. Nature Reviews Microbiology 18(11),607-621. doi: https://doi.org/10.1038/s41579-020-0412-1
[37] Longley, R., Noel, Z., Benucci, G., Chilvers, M., Trail, F. Bonito, G. (2020). Crop Management Impacts the Soybean (Glycine max) Microbiome. Frontiers in Microbiology 11,1116. doi: https://doi.org/10.3389/fmicb.2020.01116
[38] Miura, T., Sánchez, R., Castañeda, L., Godoy, K. y Barbosa, O. (2019). Shared and unique features of bacterial communities in native forest and vineyard phyllosphere. Ecology and evolution 9(6),3295-3305. doi: https://doi. org/10.1002/ece3.4949
[39] Qu, Q., Zhang, Z., Peijnenburg, W., Liu, W., Lu, T., Hu, B. y Qian, H. (2020). Rhizosphere microbiome assembly and its impact on plant growth. Journal of agricultural and food chemistry 68(18),5024-5038. doi: https://doi.org/10.1021/ acs.jafc.0c00073
[40] Sun, A., Jiao, X., Chen, Q., Wu, A., Zheng, Y., Lin, Y. y Hu, H. (2021). Microbial communities in crop phyllosphere androot endosphere are more resistant than soil microbiota to fertilization. Soil Biology and Biochemistry 153,108113.doi: https://doi.org/10.1016/j.soilbio.2020.108113
[41] Singh, U., Choudhary, A. y Sharma, S. (2020). Comparative performance of conservation agriculture vis-a-vis organic and conventional farming, in enhancing plant attributes and rhizospheric bacterial diversity in Cajanus cajan: A field study. European Journal of Soil Biology 99,103197. doi: https://doi.org/10.1016/j.ejsobi.2020.103197
[42] Lupatini, M., Korthals, G., de Hollander, M., Janssens,T. y Kuramae, E. (2017). Soil microbiome is more heterogeneous in organic than in conventional farming system. Frontiers in microbiology 7,2064. doi: https://doi.org/10.3389/ fmicb.2016.02064
[43] Zhang, J., Bei, S., Li, B., Zhang, J., Christie, P. & Li, X. (2019). Organic fertilizer, but not heavy liming, enhances banana biomass, increases soil organic carbon and modifies soil microbiota. Applied Soil Ecology 136,67-79. doi: https://doi. org/10.1016/j.apsoil.2018.12.017
[44] Zhang, M., Zhang, X., Zhang, L., Zeng, L., Liu, Y., Wang, X. y Ai, C. (2021). The stronger impact of inorganic nitrogen fertilization on soil bacterial community than organic fertilization in short-term condition. Geoderma 382,114752. doi: https://doi.org/10.1016/j.geoderma.2020.114752
[45] Peruzzi, E., Franke-Whittle, I. H., Kelderer, M., Ciavatta, C. y Insam, H. (2017). Microbial indication of soil health in apple orchards affected by replant disease. Applied Soil Ecology 119,115-127. doi: https://doi.org/10.1016/j. apsoil.2017.06.003
[46] Aparna, K., Pasha, M., Rao, D. y Krishnaraj, P. (2014). Organic amendments as ecosystem engineers: microbial, biochemical and genomic evidence of soil health improvement in a tropical arid zone field site. Ecological engineering 71,268-277. doi: https://doi.org/10.1016/j.ecoleng.2014.07.016
[47] Lupatini, M., Korthals, G., Roesch, L. y Kuramae, E. (2019). Long-term farming systems modulate multi-trophic responses. Science of the Total Environment 646,480-490. doi: https://doi.org/10.1016/j.scitotenv.2018.07.323
[48] Bengtsson, J., Ahnström, J. y Weibull, A. (2005). The effects of organic agriculture on biodiversity and abundance: a meta‐analysis. Journal of applied ecology 42(2),261-269. doi: https://doi.org/10.1111/j.1365-2664.2005.01005.x
[49] Tian, W., Wang, L., Li, Y., Zhuang, K., Li, G., Zhang, J. y Xi, Y. (2015). Responses of microbial activity, abundance, and community in wheat soil after three years of heavy fertilization with manure-based compost and inorganic nitrogen. Agriculture, Ecosystems & Environment 213,219-227. doi: https://doi.org/10.1016/j.agee.2015.08.009
[50] Tian, W., Zhang, Z., Hu, X., Tian, R., Zhang, J., Xiao, X. y Xi, Y. (2015). Short-term changes in total heavy metal concentration and bacterial community composition after replicated and heavy application of pig manure-based compost in an organic vegetable production system. Biology and Fertility of Soils, 51(5),593-603. doi: https://doi. org/10.1007/s00374-015-1005-4
[51] Fierer, N., Bradford, M. A., & Jackson, R. B. (2007). Toward an ecological classification of soil bacteria. Ecology, 88(6),1354-1364. doi: https://doi.org/10.1890/05-1839
[52] Navarrete, A., Kuramae, E., de Hollander, M., Pijl, A., van Veen, J. y Tsai, S. M. (2013). Acidobacterial community responses to agricultural management of soybean in Amazon forest soils. FEMS Microbiology Ecology 83(3),607–621. doi: https://doi.org/10.1111/1574-6941.12018
[53] Bonanomi, G., De Filippis, F., Cesarano, G., La Storia, A., Ercolini, D. y Scala, F. (2016). Organic farming induces changes in soil microbiota that affect agro-ecosystem functions. Soil Biology and Biochemistry 103,327-336. doi: https://doi. org/10.1016/j.soilbio.2016.09.005
[54] Banerjee, S., Kirkby, C., Schmutter, D., Bissett, A., Kirkegaard, J. y Richardson, A. (2016). Network analysis reveals functional redundancy and keystone taxa amongst bacterial and fungal communities during organic matter decomposition in an arable soil. Soil Biol. Biochem. 97,188–198. doi: https://doi.org/10.1016/j.soilbio.2016.03.017
[55] Kalam, S., Basu, A., Ahmad, I., Sayyed, R.., El Enshasy, H., Dailin, D. y Suriani, N. (2020). Recent understanding of soil Acidobacteria and their ecological significance: A critical review. Frontiers in Microbiology, 11,2712. doi: https://doi. org/10.3389/fmicb.2020.580024
[56] Touceda, M., Brader, G., Antonielli, L., Ravindran, V., Waldner, G., Friesl-Hanl, W. y Sessitsch, A. (2015). Combinedamendment of immobilizers and the plant growth-promoting strain Burkholderia phytofirmans PsJN favours plantgrowth and reduces heavy metal uptake. Soil Biology and Biochemistry 91,140-150. doi: https://doi.org/10.1016/j.soilbio.2015.08.038
[57] Teng, Y., Wang, X., Li, L., Li, Z., & Luo, Y. (2015). Rhizobia and their bio-partners as novel drivers for functional remediation in contaminated soils. Frontiers in plant science, 6, 32. doi: https://doi.org/10.3389/fpls.2015.00032
[58] Ma, L., Geiser, D. M., Proctor, R. H., Rooney, A. P., O’Donnell, K., Trail, F. y Kazan, K. (2013). Fusarium pathogenomics. Annual review of microbiology 67,399-416. doi: https://doi.org/10.1146/annurev-micro-092412-155650
[59] Wakelin, S., Warren, R., Harvey, P. y Ryder, M. (2004). Phosphate solubilization by Penicillium spp. closely associated with wheat roots. Biology and Fertility of Soils 40(1),36-43. doi: https://doi.org/10.1007/s00374-004-0750-6
[60] Amaresan, N., Kumar, M., Annapurna, K., Kumar, K. y Sankaranarayanan, A. (Eds.). (2020). Beneficial Microbes in Agro-Ecology:Bacteria and Fungi. Elsevier. Recuperado de: https://elsevier.com/books-and-journals