Evaluación de marcadores microsatélites (SSRs) heterólogos en Rubus niveus para estudios de diversidad genética en las Islas Galápagos
ACI Avances en Ciencias e Ingenierías
Universidad San Francisco de Quito, Ecuador
Recepción: 14 Mayo 2021
Aprobación: 09 Septiembre 2021
Resumen: Rubus niveus o mora es una especie originaria de Asia. Esta planta se ha dispersado por varios continentes por sus usos antropogénicos y sus características biológicas. La alta adaptabilidad que presenta le ha permitido establecerse en nuevos ambientes y volverse invasora, como es el caso de las Islas Galápagos, Ecuador. Desde su llegada a estas islas, la mora ha ido desplazando plantas nativas y hasta el momento ninguno de los métodos de control utilizados ha resultado efectivo. Estudiar la diversidad genética de esta planta usando marcadores moleculares podría servir para explicar el éxito de su invasión y para desarrollar medidas de control efectivas. Por lo expuesto, en el presente estudio se evaluó la viabilidad de marcadores microsatélites heterólogos para el análisis de la diversidad genética de esta especie en las Islas Galápagos. Para ello se colectaron y analizaron 68 muestras de diferentes localidades de las islas Santa Cruz, San Cristóbal, Isabela y Floreana, y 10 muestras del Ecuador continental. Se escogieron 15 marcadores microsatélites y todos amplificaron exitosamente, demostrando transferibilidad de una especie a otra. Los 15 loci resultaron monomórficos para las muestras amplificadas de las Islas Galápagos y Ecuador continental. Estos resultados no permitieron determinar la diversidad genética de la mora en las muestras estudiadas. Nuestro estudio con microsatélites y otros similares en especies del género Rubus, encontraron loci monomórficos. Por lo tanto, sugerimos llevar a cabo el secuenciamiento del genoma de la mora y así utilizar otros marcadores moleculares como los polimorfismos de nucleótido simple para dilucidar el nivel de diversidad genética de esta especie.
Palabras clave: Rubus niveus, especie invasora, Islas Galápagos, marcadores microsatélite, diversidad genética, monomorfismo.
Abstract: Rubus niveus is a species that originated in Asia. This plant has spread over several continents due to its anthropogenic uses and biological characteristics. It presents high adaptability, which has allowed it to establish in new environments and invade them, as is the case of the Galapagos Islands, Ecuador. Since it arrived in the archipelago, R. niveus has displaced native plants. Existing control methods have so far been ineffective. Understanding this plant’s genetic diversity using molecular markers could explain its invasive success, and aid in developing efficient control strategies. Therefore, in this study, we carried out a preliminary analysis of the transferability of heterologous microsatellite markers for the study of genetic diversity of R. niveus in the Galapagos Islands. For this purpose, we collected and analyzed 68 samples from different locations within Santa Cruz, San Cristóbal, Isabela, and Floreana islands, and 10 samples from continental Ecuador. We chose 15 microsatellite markers for this study, all of which amplified successfully, demonstrating transferability from one species to another. However, all 15 loci were monomorphic in every amplified sample from the Galapagos Islands and continental Ecuador; therefore, we were unable to determine the genetic diversity of R. niveus in our samples. Our research with microsatellite markers and similar studies with species in the Rubus genus found monomorphic loci. Therefore, we suggest that a better strategy would be to sequence R. niveus’ genome and explore other molecular markers such as Single Nucleotide Polymorphisms to determine the genetic diversity levels of this species. Vol. 13, nro. 2 ID: 2293
Keywords: Rubus niveus, invasive species, Galapagos Islands, microsatellite markers, genetic diversity, monomorphism.
INTRODUCCIÓN
Rubus niveus (Thunb.), comúnmente conocida como “mora” en el Ecuador, es un arbusto perenne perteneciente a la familia Rosaceae. Esta especie es originaria de Asia, específicamente de las zonas templadas del sistema de los Himalayas [1]. Su fruto es altamente apetecible por su sabor agradable y alto contenido nutricional [2], razón por la cual se comenzó a comercializar aproximadamente en la década de 1930 desde la India hacia el resto del mundo [3]. Actualmente, se encuentra distribuida en varios continentes como Asia, Oceanía, América y África [3, 4].
Las semillas de esta especie son altamente viables y tienen un desarrollo rápido [3]. Por esta razón, la mora se puede encontrar en varios ambientes, incluyendo bordes de carreteras, campos abiertos, bosques no perturbados [5] y áreas urbanas [6]. Además, tiene una alta capacidad competitiva por recursos naturales, rápida madurez y múltiples modos de reproducción [7,8]. Estas características han convertido a la mora en un gran problema en distintos archipiélagos [3], tales como Hawái y Galápagos, donde es considerada como una especie invasora [1].
El archipiélago de Galápagos es una región que presenta un alto grado de endemismo en varios grupos taxonómicos [9]. Esta región es vulnerable a plantas invasoras por su clima variado, su tipo de suelo y sus distintos rangos altitudinales [10]. Varios estudios indican que la amplia cobertura que existe de la mora en las Islas Galápagos se debe a su propagación vegetativa y la fácil dispersión de sus semillas [1,3,11]. Es así como Rubus niveus ha proliferado en el archipiélago y se encuentra distribuida en varias islas como San Cristóbal, Santa Cruz, Floreana, Santiago e Isabela [1], desplazando a especies nativas como Miconia robinsoniana, Cyathea weatherbyana y Scalesia pedunculata [1,4]. Desde una perspectiva de manejo y para controlar el impacto que puede tener esta especie en la ecología de las Islas Galápagos, se ha tratado de mantener la cobertura de la mora por debajo del 60% [12]. Para lograr este objetivo, se han implementado distintos métodos que combinan, tanto el control manual como el químico [12,13]. Sin embargo, hasta el momento, ninguno de estos métodos ha sido efectivo para controlar la población de la mora en las Islas Galápagos [4].
La genética y la ecología de poblaciones están vinculadas con los eventos de invasiones biológicas, ya que a menudo las especies invasoras enfrentan importantes desafíos de adaptación a nuevos entornos y condiciones ecológicas, que pueden ser distintas a las encontradas en su rango de distribución nativo [14]. En este contexto, el uso de marcadores moleculares permitiría estudiar procesos ecológicos, como la colonización, dispersión y las interacciones ecológicas dentro de una comunidad [14]. Además, estos marcadores pueden proveer evidencia de la ocurrencia de múltiples introducciones, proporcionar información sobre el patrón espacial de invasión, permitir identificar la población o poblaciones de origen, e identificar la variación genética perdida por cuellos de botella durante sucesos de colonización [15]. En estudios de diversidad genética y estructura poblacional, el análisis de marcadores moleculares contribuiría a entender la dinámica poblacional de Rubus niveus. Además, permitirían inferir los posibles eventos de colonización en las diferentes islas y se podrían plantear alternativas de control y manejo adecuadas para esta especie [1,16].
Se han reportado algunos estudios sobre la diversidad genética de especies del género Rubus. En R. idaeus se utilizaron Random Amplified Polymorphic DNA (RAPDs) para detectar diversidad genética, mientras que en R. glaucus se utilizaron Amplified Fragment Length Polymorphism (AFLPs) con el mismo propósito [17,18]. Estos dos tipos de marcadores también han sido empleados para analizar el grado de similitud y diversidad genética de varias especies de Rubus [17-21]. Por otra parte, los marcadores Inter Simple Sequence Repeats (ISSRs) y microsatélites o Simple Sequence Repeats (SSRs), han sido utilizados para la caracterización de germoplasma y programas de fitomejoramiento en R. chamaemorus, R. coreanus, R. occidentalis y R. glaucus [22-25]. ISSRs, AFLPs y espaciadores intergénicos fueron empleados en R. phoenicolasius, R. takesimensis y R. aceifolius para el estudio de la diversidad genética de especies de Rubus invasoras de ecosistemas forestales e insulares [26-28]. A pesar de contar con toda esta información, hasta el momento no se ha analizado a nivel molecular, la diversidad genética de R. niveus en las Islas Galápagos.
El estudio de la diversidad genética de una especie invasora puede servir para explicar su éxito ecológico, la capacidad de las poblaciones para adaptarse a condiciones ambientales nuevas o cambiantes y para diseñar planes de manejo efectivos [7,15,16]. Los SSRs han demostrado ser polimórficos y transferibles entre múltiples especies de Rubus, por lo que han sido útiles para el estudio de la diversidad genética, estructura poblacional y relaciones evolutivas [23,25,29,30]. Por esta razón, el objetivo de este estudio fue evaluar la viabilidad de uso de marcadores SSRs heterólogos para el análisis preliminar de la diversidad genética de esta especie en las Islas Galápagos.
MATERIALES Y MÉTODOS
Recolección de muestras
Se recolectaron 68 muestras de hojas jóvenes y sanas de Rubus niveus en cuatro islas: San Cristóbal (SCR), Santa Cruz (STX), Isabela (ISA) y Floreana (FLO) (Tabla 1). Se tomaron de cinco a siete foliolos por planta. Todas las muestras fueron recolectadas al borde de caminos, carreteras y vías principales, excepto las muestras de Floreana que fueron recolectadas en el área del Parque Nacional Galápagos (PNG), al igual que cuatro muestras en San Cristóbal (El Junco) y tres muestras en Santa Cruz (Los Gemelos) (Fig. 1). Las muestras fueron almacenadas en bolsas plásticas con cierre hermético que contenían gel de sílice. Posteriormente, se trasladaron al Galapagos Science Center (GSC) en San Cristóbal y se mantuvieron a temperatura ambiente hasta ser trasladadas al Laboratorio de Biotecnología Vegetal de la Universidad San Francisco de Quito (USFQ) en Quito, Ecuador.
| Localidad | Código | N° muestras recolectadas | |
| Isla San Cristóbal | SCR | 18 | |
| Galápagos | Isla Santa Cruz | STX | 18 |
| Isla Isabela | ISA | 18 | |
| Isla Floreana | FLO | 14 | |
| Av. Intervalles (Pichincha) | NT | 1 | |
| Cumbayá (Pichincha) | CUM | 1 | |
| Ecuador | Valle de los Chillos (Pichincha) | VCH | 2 |
| continental | Puembo (Pichincha) | PUE | 2 |
| Mindo (Pichincha) | MIN | 2 | |
| Riobamba (Chimborazo) | RIO | 2 | |
Número de muestras de Rubus niveus recolectadas por localidad en las Islas Galápagos y el Ecuador continental, y códigos utilizados para identificar localidades.
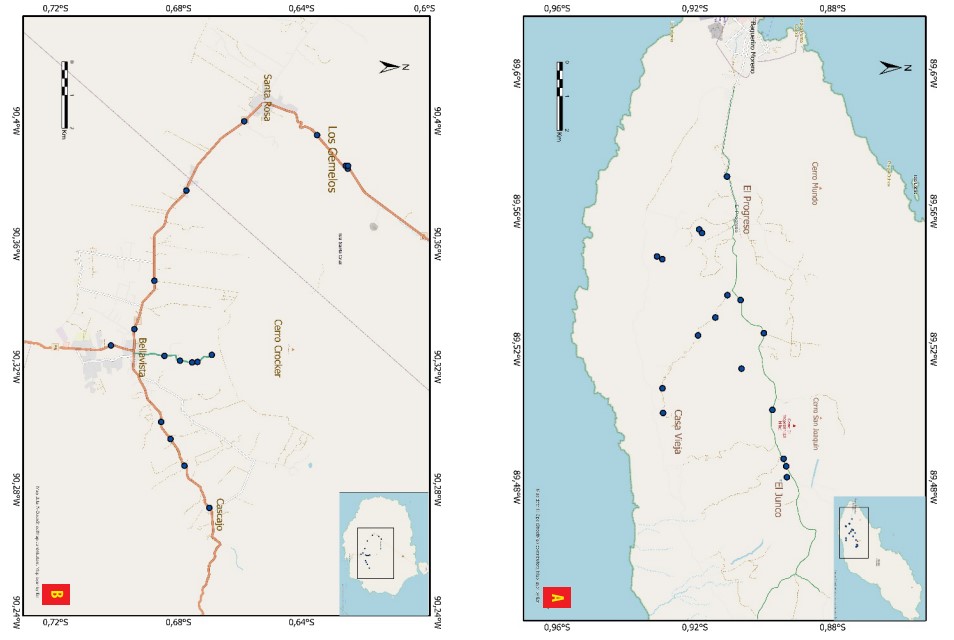
Figura 1
Mapa de los sitios de muestreo de hojas de Rubus niveus en cuatro islas del archipiélago de Galápagos. (1A) Isla San Cristóbal, (1B) Isla Santa Cruz, (1C) Isla Isabela, (1D) Isla Floreana. Mapa realizado con ArcGIS Pro 2.7.0 Esri Inc. (2020).
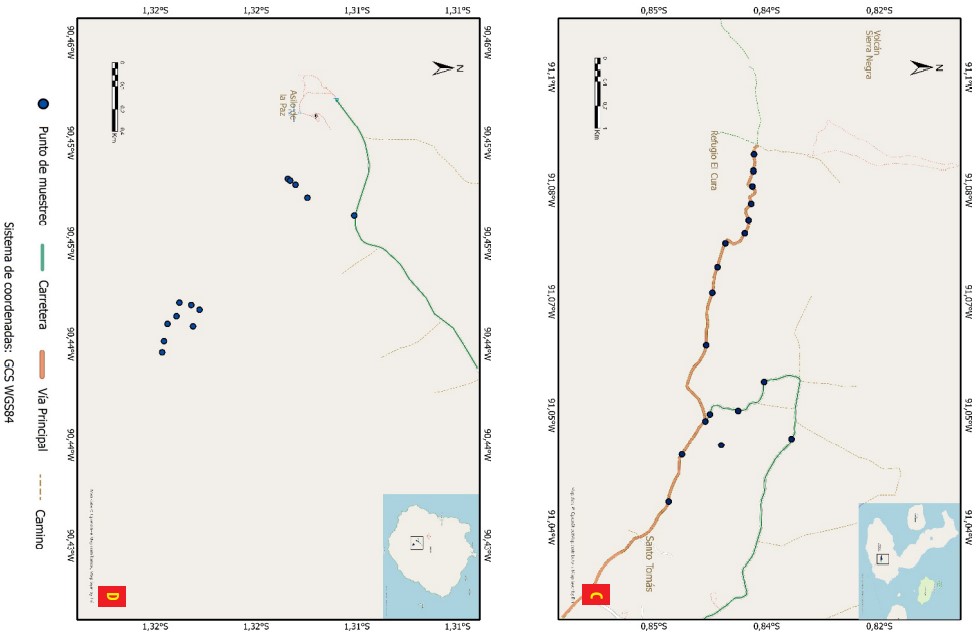
Figura 1 (continuación)
Por otra parte, se recolectaron 10 muestras de hojas sanas de Rubus niveus dentro de Ecuador continental (Tabla 1). Estas fueron recolectadas al borde de las carreteras (Fig. 2) endone y almacenadas en bolsas plásticas con cierre hermético que contenían gel de sílice, para ser trasladadas al Laboratorio de Biotecnología Vegetal de la Universidad San Francisco de Quito (USFQ) en Quito, Ecuador.
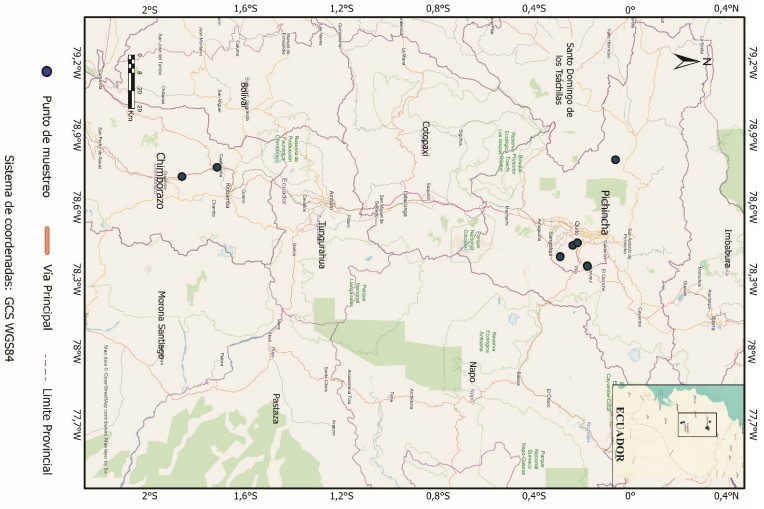
Figura 2
Mapa de los sitios de muestreo de hojas de Rubus niveus en dos provincias de Ecuador continental: Pichincha y Chimborazo. Mapa realizado con ArcGIS Pro 2.7.0 Esri Inc. (2020).
La recolección de muestras y las actividades de esta investigación se realizaron bajo el Contrato Marco MAE-DNB-CM-2016-0041, firmado entre la USFQ y el Ministerio del Ambiente del Ecuador.
Extracción ADN
El ADN se extrajo siguiendo el protocolo de CTAB (Cetrimonium Bromide) para aislamiento de ADN vegetal de alto peso molecular [31]. El gel fue revelado mediante el software Image Lab™ del sistema Gel Doc XR+ (Bio-Rad Laboratories, Inc.). El ADN extraído se cuantificó por medio de un espectrofotómetro de UV visible Nanodrop 2000TM de Thermo ScientificTM. La calidad del ADN genómico se confirmó mediante un gel de agarosa al 1% (p/p) teñido con SYBR Safe (Invitrogen). Las muestras de ADN se diluyeron en agua ultrapura a concentraciones de aproximadamente 20 ng/gl.
Elección de marcadores SSRs
Se utilizaron 15 marcadores SSRs diseñados para las especies Rubus idaeus, R. glaucus, R. alceifolius y el híbrido Marion blackberry. Los marcadores SSRs escogidos han reportado previamente un alto polimorfismo y alta transferibilidad [25,32,33]. Con el fin de facilitar el trabajo en laboratorio, se renombró los 15 marcadores SSRs con un nuevo código de tres caracteres (R01- R15). El detalle de los marcadores SSRs escogidos se indica en la Tabla 2.
| Locus | Código SSRs | Especie | Motivo | Tamaño esperado (pb) | Referencia | ||
| Rubus116a | R01 | R. idaeus | (CT)12(T)10 | 299 | [30] | ||
| Rg-A12-1 | R02 | R. glaucus | (GA)23 | 387 - 432 | [30] | ||
| RhM003 | R03 | Rubus hybrid, Marion blackberry | (TG)10 | 173 - 264 | [18,25] | ||
| Rubus16a | R04 | R. idaeus | (AT)8(GT)11 | 169 | |||
| Rubus 76b | R05 | R. idaeus | (CT)5(CT)4 | 190 - 210 | [25,30] | ||
| RiM019 | R06 | Rubus hybrid, Marion blackberry | (AG)12 | 146 - 196 | [25,32] | ||
| Rubleaf97 | R07 | R. idaeus | (CTT)-(CCT)9 | 205 | [33] | ||
| RhM011 | R08 | Rubus hybrid, Marion blackberry | (TC)18 | 252 - 346 | [25,32] | ||
| mRaCIRRIH3 | R09 | R. alceifolius | (GT)15(GA)17 | 160 - 226 | [25] |
Información de los marcadores SSRs heterólogos escogidos para la amplificación en Rubus niveus. En negrita se encuentran los SSRs compuestos.
| Locus | Código SSRs | Especie | Motivo | Tamaño esperado (pb) | Referencia | |
| Rubus126b | R10 | R. idaeus | (CT)31(CA)22 | 150 - 201 | [33] | |
| Rubus98d | R11 | R. idaeus | (GAA)5(GA)10 | 198 | [30] | |
| Rubus 259f | R12 | R. idaeus | (CT)4(AG)8 | 265 | [25] | |
| RhM021 | R13 | R. idaeus | (TC)6 | 282 | [25,30] | |
| Rubus105b | R14 | R. idaeus | (AG)8 | 165 - 173 | [25,30] | |
| Rubus 275a | R15 | R. idaeus | (AG)27 | 114 - 145 | [33] | |
Estandarización primers heterólogos
Para la amplificación de los marcadores SSRs, se utilizó la metodología descrita por [34], la misma que consiste en el uso de tres primers: un primer universal marcado con un fluoróforo (6-FAM, VIC, NED, PET), un primer forward modificado con una secuencia corta adicional complementaria a la del primer universal y un primer reverse [34]. Este protocolo ha sido estandarizado previamente en el Laboratorio de Biotecnología Vegetal de la USFQ.
Para la amplificación de 15 SSRs en R. niveus, las concentraciones de los reactivos de la PCR fueron: 1X de Buffer (Invitrogen), 0,04 mg/ml de BSA, 0,32 mM de dNTPs (Invitrogen), 0,5 pM de primer reverse, 0,15 pM de primer forward, 0,5 pM del primer forward universal marcado con un fluoróforo y 1 U de Taq platinum. En el caso de MgCl
2, las concentraciones variaron de acuerdo al primer utilizado. La concentración final de ADN utilizada fue de 20 ng/ul para la mayoría de los primers, excepto para los primers R04 y R12 donde se utilizaron 40 ng/ul (Tabla 3). El volumen final de la reacción fue de 25 pl. El programa de termociclado consistió en una desnaturalización inicial a 94°C por 5 min, seguido de 35 ciclos de desnaturalización a 94°C por 30 s, annealing durante 45 s según la temperatura estandarizada para cada primer (Tabla 3) y una extensión a 72°C por 45 s. La etapa final fue una extensión a 72°C durante 10 min. Finalmente, los amplicones obtenidos se visualizaron en un gel de agarosa al 1,5% p/p, con el fotodocumentador Gel Doc XR+ (Bio-Rad Laboratories, Inc.).
| Código SSRs | Temperatura de annealing (°C) | MgCl2 (mM) | ADN (ng/ Ml) | Tamaño de amplicón (pb) |
| R01 | 61 | 1,6 | 20 | 196 |
| R02 | 53 | 2,0 | 20 | 179 |
| R03 | 51 | 2,0 | 20 | 215 |
| R04 | 57 | 2,5 | 40 | 148 |
Temperatura de annealing y concentración final de ADN y MgCl2 para cada marcador SSR. Se detalla también el tamaño amplificado en Rubus niveus para cada marcador.
| Código SSRs | Temperatura de annealing (°C) | MgCl2 (mM) | ADN (ng/ Ml) | Tamaño de amplicón (pb) | |
| R05 | 51 | 1,6 | 20 | 208 | |
| R06 | 58 | 2,0 | 20 | 210 | |
| R07 | 53 | 2,0 | 20 | 229 | |
| R08 | 56 | 2,0 | 20 | 295 | |
| R09 | 53 | 2,0 | 20 | 163 | |
| R10 | 53 | 2,0 | 20 | 164 | |
| R11 | 51 | 2,0 | 20 | 194 | |
| R12 | 59 | 2,5 | 40 | 259 | |
| R13 | 55 | 2,0 | 20 | 302 | |
| R14 | 58 | 1,6 | 20 | 165 | |
| R15 | 55 | 2,0 | 20 | 148 |
Genotipado
Se enviaron tres series de placas MicroAmpTM Optical de Applied BiosystemsTM de los amplicones obtenidos de las muestras de mora analizadas a Macrogen Inc., Corea. El primer envío fue de las 68 muestras de las Islas Galápagos amplificadas con los primers R01, R02, R03 y R04 (Tabla 2). En una segunda placa, se enviaron los 11 primers restantes (R05-R15) (Tabla 2) para un número de muestras representativo de las Islas Galápagos (15 muestras): tres de Floreana (FLO001, FLO002 y FLO013), cuatro de Isabela (ISA001, ISA005, ISA011 e ISA018), cuatro de Santa Cruz (STX005, STX011, STX015 y STX021) y cuatro de San Cristóbal (SCR001, SCR005, SCR024 y SCR029). Finalmente, el tercer envío fue de las 10 muestras del Ecuador continental amplificadas con los 15 marcadores SSRs (Datos S1).
El genotipado de los SSRs se realizó mediante una electroforesis capilar utilizando un secuenciador Applied Biosystems ABI 3130xl y el marcador de referencia de tamaño 500 LIZ (Humanizing Genomics Macrogen). Los resultados de Macrogen, Inc. se analizaron con el software GeneMarker de SoftGenetics LLC. (2012). Con este programa se identificaron los alelos de cada locus en base a su tamaño y al patrón de fluorescencia de mayor intensidad en el electroferograma. Se usaron como referencia el tamaño y el motivo de los loci, reportados en la literatura (Tabla 2).
RESULTADOS
Extracción de ADN
La extracción de ADN genómico de las 78 muestras de mora provenientes de las Islas Galápagos y del Ecuador continental fue exitosa. La concentración promedio de ADN genómico fue de 1.316,6 ng/gl. Los índices de absorbancia A260/A280 y A260/A230 promedio fueron de 1,9 y 1,7 respectivamente, lo que indica que se obtuvo un ADN de buena calidad [35,36]. Se obtuvo una banda de alto peso molecular en el gel de agarosa, lo que confirmó la buena calidad del ADN (Fig. 3).
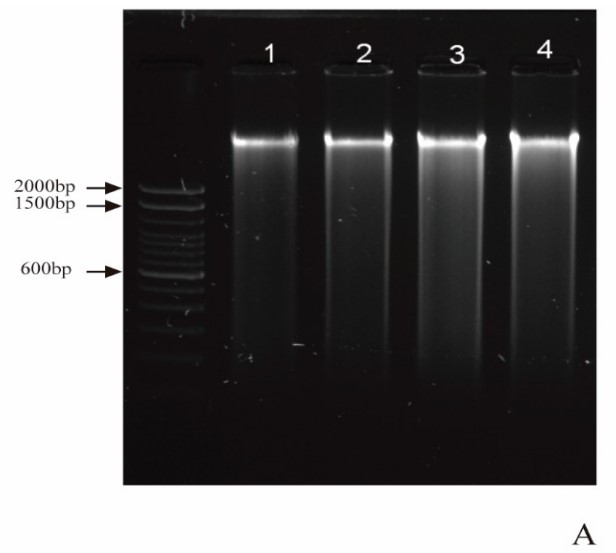
Figura 3
Calidad del ADN genómico extraído de hojas de Rubus niveus visto en un gel de agarosa al 1% (p/p).
Estandarización de primers heterólogos y amplificación de SSRs
Los 15 marcadores SSRs amplificaron exitosamente en las muestras colectadas en las Islas Galápagos y en el Ecuador continental. Trece marcadores mostraron una banda clara y solo los loci R07 y R10 presentaron bandas tenues. Un ejemplo de la amplificación de uno de los loci de microsatélite probados puede observarse en la Fig. 4.
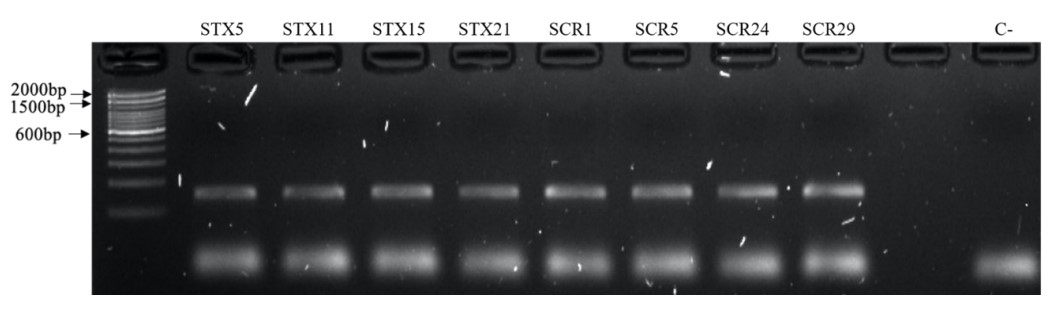
Figura 4
Electroforesis en gel de agarosa al 1,5% (p/p) de los productos de amplificación con el marcador SSR R14, en ocho muestras de Rubus niveus de las islas Santa Cruz (STX) y San Cristóbal (SCR).
Genotipado
El tamaño de los alelos encontrados para cada locus tuvo concordancia con el reportado en la literatura (Tabla 3), tanto para las muestras de las Islas Galápagos como para las del Ecuador continental. Las 68 muestras de las Islas Galápagos fueron amplificadas y genotipadas con los primers R01-R04 y resultaron monomórficos, ya que se observó un solo alelo para todas las muestras con estos marcadores. Los SSRs R05-R15 que se genotiparon con 15 muestras representativas de las Islas Galápagos también resultaron monomórficos. Finalmente, las 10 muestras del Ecuador continental se amplificaron y se genotiparon con los primers R01–R015, obteniéndose nuevamente un solo alelo en todas las muestras estudiadas. Los electroferogramas representativos para cada locus microsatélite se observan en la Fig. 5. Un ejemplo del monomorfismo observado para los loci se observa en la Fig. 6, en donde se han sobrelapado los electroferogramas obtenidos para las muestras de Ecuador continental y se distingue únicamente un alelo para cada locus en las muestras analizadas. Debido a que todos los loci analizados resultaron ser monomórficos, no fue posible realizar análisis estadísticos de la diversidad genética.
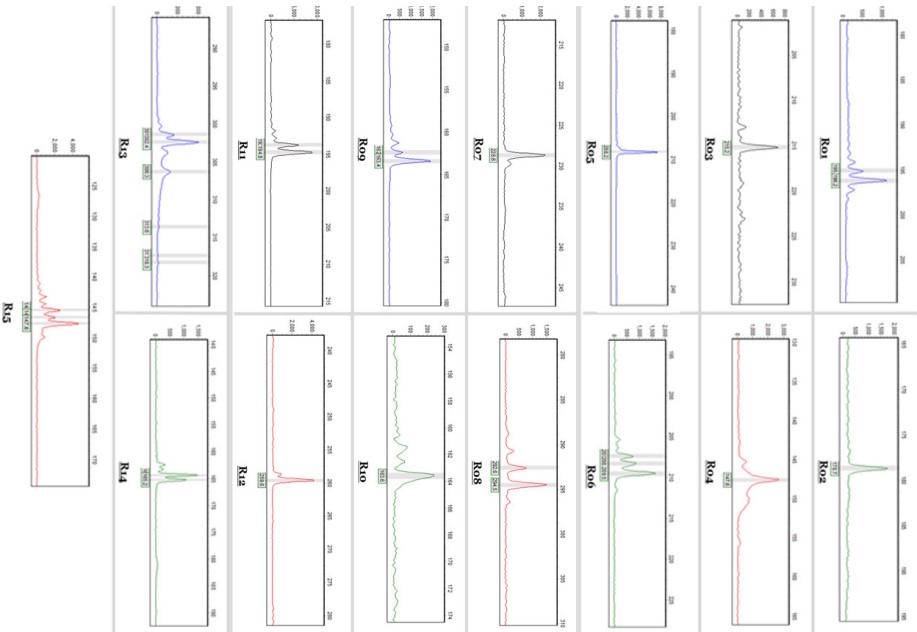
Figura 5
Electroferogramas obtenidos de los 15 loci SSRs utilizados en este estudio.
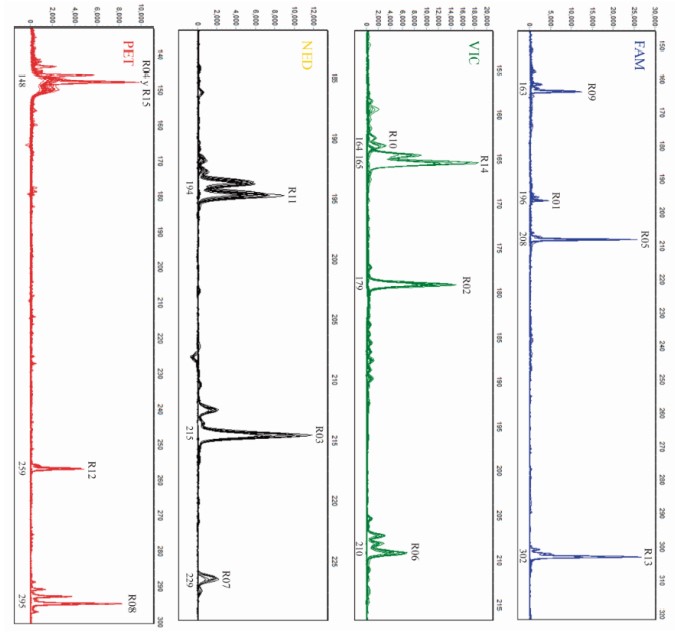
Figura 6
Superposición de los electroferogramas de siete muestras de Ecuador continental, amplificadas con cada uno de los marcadores SSRs utilizados en este estudio.
DISCUSIÓN
La concentración y calidad del ADN fueron adecuadas para llevar a cabo los análisis moleculares. El valor A260/A280 se encontró dentro del rango que indica un ADN libre de contaminantes como proteínas y polisacáridos. La absorbancia en el rango A260/A230 fue cercana al valor ideal (~1,8) [35].
Los 15 marcadores SSRs probados amplificaron y el tamaño de estas bandas tuvo concordancia con el reportado en la literatura [25,32,33,37]. Por otra parte, el genotipado reveló alelos monomórficos en cada uno de los 15 loci analizados, tanto en las poblaciones de las Islas Galápagos (68 muestras) como en la de Ecuador continental (10 muestras), siendo este resultado totalmente contrario al previsto, pues se esperaba encontrar polimorfismos como los reportados en varios estudios de los que se tomaron los primers utilizados en esta investigación [25,32,33,37]. Adicionalmente, se esperaba encontrar individuos heterocigotos debido a que Rubus niveus es una especie diploide [38,39]. Los resultados obtenidos con los marcadores SSRs heterólogos utilizados no fueron informativos y, por lo tanto, no se pudieron realizar los análisis de diversidad genética previstos. Una posibilidad para superar esta limitación encontrada podría ser el desarrollo de marcadores SSRs específicos para R. niveus.
Es importante señalar que esta no es la única investigación en la que se han reportado alelos monomórficos en el género Rubus. En el estudio de [23] se analizaron 97 SSRs para evaluar su transferibilidad en seis especies de Rubus, y solo 29 fueron polimórficos. En dos estudios que utilizaron los primers R04, R08 y R13, que también fueron utilizados en nuestra investigación, los autores indican que el genotipado con estos primers de algunas especies del género Rubus, como R. occidentalis, pueden dar como resultado alelos monomórficos [20,40]. Esta información es relevante, ya que R. niveus y R. occidentalis pertenecen al mismo subgénero que R. idaeus (Idaeobatus), la especie para la que fueron diseñados [41]. Esto sugiere que, incluso en especies estrechamente relacionadas con la especie de donde provienen los primers, se pueden encontrar loci monomórficos. Estos resultados apoyarían la idea que los SSRs no son marcadores genéticos muy informativos en ciertas especies del género Rubus, entre ellas R. niveus.
Es importante tomar en cuenta que, en los SSRs, obtener alelos de un mismo tamaño no siempre significa que la secuencia de ADN de los alelos sea la misma. En ocasiones, el tamaño de los SSRs no resulta de la repetición perfecta de sus motivos. También se forman por la repetición de más de un motivo y con secuencias cortas intercaladas entre motivos [42]. Es así que se ha reportado que los SSRs tienen cierta cantidad de homoplasia molecular, es decir, que presentan alelos con un mismo tamaño, pero con secuencia distinta [43]. Esto es común sobre todo en SSRs compuestos, es decir, formados por más de un motivo [43,44]. Ocho de los 15 SSRs utilizados en el presente estudio son compuestos (Tabla 2) por lo que los resultados obtenidos podrían estar relacionados con esta característica.
Una alternativa para el análisis de homoplasia molecular en SSRs es la metodología High-Resolution Melting (HRM) [40]. Esta técnica consiste en añadir un paso de desnaturalización luego de la extensión final en el programa de termociclado de la PCR. Se usa un tinte intercalante para visualizar curvas de fluorescencia generadas por diferentes temperaturas de melting (Tm) de los amplicones, pues diferentes Tm podrían indicar diferencias en las secuencias [45]. Esta técnica ha sido usada previamente en el género Rubus con la especie R. occidentalis cuando se encontraron marcadores SSRs monomórficos [40]. En el estudio citado, se pudieron detectar diferentes Tm en las secuencias con la técnica HRM en algunos loci. Luego de secuenciar estos alelos se pudo concluir que, efectivamente, existen diferencias en las secuencias de los alelos del mismo tamaño [40]. Esto indicaría que la técnica del HRM podría dilucidar que, a pesar de encontrar un mismo tamaño de alelo, la secuencia no es necesariamente la misma.
Sin embargo, considerando la tecnología disponible actualmente, una mejor estrategia podría ser la secuenciación del genoma (WGS, Whole Genome Sequencing) de Rubus niveus, lo que aportaría con información amplia de esta especie y un número alto de marcadores moleculares, como se evidencia en otras especies de Rubus de importancia económica que han sido secuenciadas [41]. La técnica de WGS es cada vez menos costosa y más accesible, además de ser la herramienta más completa para estudios de variabilidad y diversidad genética [46]. El secuenciamiento ha servido para diseñar nuevos marcadores, como los polimorfismos de un solo nucleótido (SNPs), que ya han presentado buenos resultados en la determinación de variabilidad genética en otras especies del género Rubus [47-49]. Por lo tanto, esta alternativa podría ser eficiente para determinar la diversidad genética de R. niveus en las Islas Galápagos.
Conocer sobre la diversidad genética de una especie invasora como se mencionó previamente puede ser una herramienta para explorar posibles estrategias de control biológico como se ha planteado para otras especies invasoras como Rubus alceifolius, Macfadyena unguis-cati y Jatropha gossypiifolia [16,50]. Una diversidad genética baja en una población invasora podría sugerir, por ejemplo, que un agente de control biológico eficaz para un individuo de la especie, probablemente lo será para toda la población [51]. Encontrar los mismos haplotipos entre poblaciones invasoras y poblaciones nativas, puede dar indicios de cómo se puede controlar la población invasora, por ejemplo, utilizando herbívoros de la región nativa que afecten estos haplotipos [16]. Sin embargo, para poder determinar la diversidad genética se debe primero encontrar el marcador molecular ideal. Por esto, para el caso de la mora en las Islas Galápagos, en el futuro se espera tener acceso a marcadores más informativos y así poder contribuir al manejo de esta especie invasora.
AGRADECIMIENTOS
Los autores agradecen a los miembros del Laboratorio de Biotecnología Vegetal por su apoyo a la realización de esta investigación. También agradecemos a la asistente de investigación Karol Molina, que forma parte del Laboratorio de Biotecnología Agrícola, por su colaboración en la recolección de muestras. Esta investigación es parte del proyecto “Análisis de la diversidad genética de la mora en las Islas Galápagos”, liderado por María de Lourdes Torres y Antonio Leon-Reyes y que fue financiado por la Universidad San Francisco de Quito (USFQ) a través de los “Fondos COCIBA”.
CONTRIBUCIONES DE LOS AUTORES
María de Lourdes Torres y Antonio Leon-Reyes concibieron la investigación; María de Lourdes Torres y Estefanía Rojas diseñaron la metodología y curación de datos; Noelia Barriga, Pablo Alarcón, María P. Erazo-García, Mayra Ortega, Antonio Leon-Reyes se encargaron de la adquisición, análisis o interpretación de los datos, incluyendo las labores de campo, laboratorio, experimentales, estadísticas, o el soporte técnico; María de Lourdes Torres, Gabriela Pozo y Estefanía Rojas supervisaron y fueron las tutoras; María de Lourdes Torres, Gabriela Pozo y María P. Erazo-García validaron y verificaron los resultados; Pablo Alarcón, Gabriela Pozo, María P. Erazo-García, Mayra Ortega, Estefanía Rojas, Noelia Barriga redactaron el manuscrito; María de Lourdes Torres y Antonio Leon-Reyes realizaron la revisión crítica del contenido intelectual del manuscrito; Pablo Alarcón, María P. Erazo-García, Noelia Barriga, Mayra Ortega se encargaron de la producción de tablas figuras y material complementario
CONFLICTO DE INTERÉS
Los autores declaran que no existen conflictos de intereses en el presente trabajo.
Referencias
Quinton St., J. M., Fay, M. F., Ingrouille, M., & Faull, J. (2011). Characterisation of Rubus niveus: A prerequisite to its biological control in oceanic islands. Biocontrol Science and Technology, 21 (6), 733-752. doi: https://doi.org/10.1080/09583157.2011.570429
Caidan, R., Cairang, L., & Yourui, S. (2013). Simultaneous analysis of fatty acids in Rubus niveus Thunb. Fruits by HPLC- MS/MS. Asian Journal of Chemistry, 25(4), 1866-1870. doi: https://doi.org/10.14233/ajchem.2013.13204
Centre for Agricultural Bioscience International. (2019). Rubus niveus (Mysore raspberry). Recuperado de https://web.archive.org/web/20210506091150/https://www.cabi.org/isc/datasheet/107939
Global Invasive Species Database. (2021). Species profile: Rubus niveus. Recuperado de http://www.iucngisd.org/gisd/species.php?sc=1232
BIISC. (2013). Family: Rubus glaucus Mysore Raspberry Rubus niveus Thimbleberry Rubus rosifolius Koster’s Curse Clidemia hirta Glory Bush Tibouchina herbacea Lasiandra Tibouchina urvilleana. Article. Recuperado de: https://web.archive.org/web/20210514222533/http://www.hear.org/operationmiconia/BIISC_ WEEDS_01-15.pdf
Starr, F., Starr, K., & Loope, L. (2003). Rubus niveus f. a Hill or mysore raspberry Rosaceae. Recuperado de http://www.hear.org/PIER/pdf/pohreports/rubus_niveus_f_a.pdf
Renteria, J. L., Gardener, M. R., Panetta, F. D., & Crawley, M. J. (2012). Management of the Invasive Hill Raspberry (Rubus niveus) on Santiago Island, Galapagos: Eradication or Indefinite Control? Invasive Plant Science and Management, 5(1), 37-46. doi: https://doi.org/10.1614/ipsm-d-11-00043.1
Rentería, J. L., Gardener, M. R., Panetta, F. D., Atkinson, R., & Crawley, M. J. (2012). Possible Impacts of the Invasive Plant Rubus niveus on the Native Vegetation of the Scalesia Forest in the Galapagos Islands. PLoS ONE, 7(10), 1-9. doi: https://doi.org/10.1371/journal.pone.0048106
Malanson, G., & Walsh, S. (2013). A Geographical Approach to Optimization of Response to Invasive Species. En S. Walsh & C. Mena (Eds.), Social and Ecological Interactions in the Galapagos Islands (pp.199-215). Springer Science+Business Media, LLC 2013
Moity, N & Rivas, G. (2018). Ecosistemas. En P. Araujo, H. Arnal, B. Delgado, P. Díaz, A. Izurieta, G. Jiménez-Uzcátegui, J. R. Marín, N. Moity, J. Ramírez, M. Schuiteman (Eds.), Atlas de Galápagos, Ecuador: Especies Nativas e Invasoras (pp. 36). Quito: Fundación Charles Darwin y WWF-Ecuador.
Lawesson, J., & Ortiz, L. (1994). Plantas introducidas en las Islas Galápagos. En A. Carrasco & H. Valdebenito (Eds.), Investigación Botánica y Manejo en Galápagos: Memorias (Versión en Español) Taller sobre Investigación Botánica y Manejo en Galápagos Abril 11-18 de 1987 (pp. 224-235). Quito: USAID.
Jäger, H., Buchholz, S., Cimadom, A., Tebbich, S., Rodriguez, J., Barrera, D., . Causton, C. E. (2017). Restoration of the blackberry-invaded Scalesia forest: Impacts on the vegetation, invertebrates, and birds. Galapagos Report 2015-2016, 142-148.
Ministerio del Ambiente. (2009). Términos de referencia para la prestación de servicios para realizar monitoreo, delimitación y control de mora (Rubus niveus) en la isla Santiago y la isla Floreana. Santa Cruz, Ecuador.
Lawson, L. J., Estoup, A., Evans, D. M.,Thomas, C. E., Lombaert, E., Facon, B., .. Roy, H. E. (2011). Ecological genetics of invasive alien species. BioControl, 56(4), 409-428. doi: https://doi.org/10.1007/s10526-011-9386-2
Sakai, A. K., Allendorf, F. W., Holt, J. S., Lodge, D. M., Molofsky, J., With, K. A., ... Weller, S. G. (2001). The Population Biology of Invasive Species. Annual Review of Ecology and Systematics, 32(1), 305-332. doi: https://doi.org/10.1146/annurev.ecolsys.32.081501.114037
Prentis, P. J., Sigg, D. P., Raghu, S., Dhileepan, K., Pavasovic, A., & Lowe, A. J. (2009). Understanding invasion history: Genetic structure and diversity of two globally invasive plants and implications for their management. Diversity and Distributions, 15(5), 822-830. doi: https://doi.org/10.1111/j.1472-4642.2009.00592.x
Graham, J., Squire, G. R., Marshall, B., & Harrison, R. E. (2003). Spatially dependent genetic diversity within and between colonies of wild raspberry Rubus idaeus detected using RAPD markers. Molecular Ecology, 6(11), 1001-1008. doi: https://doi.org/10.1046/j.1365-294X.1997.00272.x
Garrido, P., Morillo, E., & Vásquez-Castillo, W. (2020). Genetic diversity of the Andean blackberry (Rubus glaucus Benth.) in Ecuador assessed by AFLP markers. Plant Genetic Resources: Characterisation and Utilisation, 18(4), 1-8. doi: https://doi.org/10.1017/S1479262120000283
Miyashita, T., Kunitake, H., Yotsukura, N., & Hoshino, Y. (2015). Assessment of genetic relationships among cultivated and wild Rubus accessions using AFLP markers. Scientia Horticulturae, 193, 165-173. doi: https://doi.org/10.1016/j.scienta.2015.07.004
Marulanda, M. L., López, A. M., & Aguilar, S. B. (2007). Genetic diversity of wild and cultivated Rubus species in Colombia using AFLP and SSR markers. Crop Breeding and Applied Biotechnology, 7, 242-252.
Graham, J., & McNicol, R. J. (1995). An examination of the ability of RAPD markers to determine the relationships within and between Rubus species. Theoretical and Applied Genetics, 90(7-8), 1128-1132. doi: https://doi.org/10.1007/BF00222932
Debnath, S. C. (2007). Inter-simple sequence repeat (ISSR)-PCR analysis to assess genetic diversity in a collection of wild cloudberry (Rubus chamaemorus L.) clones. Journal of Horticultural Science and Biotechnology, 82(5), 727-732. doi: https://doi.org/10.1080/14620316.2007.11512297
Lee, G. A., Song, J. Y., Choi, H. R., Chung, J. W., Jeon, Y. A., Lee, J. R., ... Lee, M. C. (2015). Novel microsatellite markers acquired from Rubus coreanus miq. and cross-amplification in other Rubus species. Molecules, 20(4), 6432-6442. doi: https://doi.org/10.3390/molecules20046432
Dossett, M., Bassil, N. V., Lewers, K. S., & Finn, C. E. (2012). Genetic diversity in wild and cultivated black raspberry (Rubus occidentalis L.) evaluated by simple sequence repeat markers. Genetic Resources and Crop Evolution, 59(8), 1849-1865. doi: https://doi.org/10.1007/s10722-012-9808-8
Marulanda, M., López, A. M., & Uribe, M. (2012). Molecular characterization of the Andean blackberry, Rubus glaucus, using SSR markers. Genetics and molecular research: GMR, 11(1), 322-331. doi: https://doi.org/10.4238/2012. February.10.3
Innis, A. F., Forseth, I. N., Whigham, D. F., & McCormick, M. K. (2011). Genetic diversity in the invasive Rubus phoenicolasius as compared to the native Rubus argutus using inter-simple sequence repeat (ISSR) markers. Biological Invasions, 13(8), 1735-1738. doi: https://doi.org/10.1007/s10530-011-0012-0
Yang, J., Pak, J.-H., Maki, M., & Kim, S.-C. (2019). Multiple origins and the population genetic structure of Rubus takesimensis (Rosaceae) on Ulleung Island: Implications for the genetic consequences of anagenetic speciation. PLoS ONE, 14(9), e0222707. doi: https://doi.org/10.1371/journal.pone.0222707
Amsellem, L., Dutech, C., & Billotte, N. (2001). Isolation and characterization of polymorphic microsatellite loci in Rubus alceifolius Poir. (Rosaceae), an invasive weed in la Réunion island. Molecular Ecology Notes, 1(1-2), 33-35. doi: https://doi.org/10.1046/j.1471-8278.2000.00013.x
Lee, K. J., Lee, G.-A., Kang, H.-K., Lee, J.-R., Raveendar, S., Shin, M.-J., ... Ma, K.-H. (2016). Genetic Diversity and Population Structure of Rubus Accessions Using Simple Sequence Repeat Markers. Plant Breeding and Biotechnology, 4(3), 345-351. doi: https://doi.org/10.9787/pbb.2016.4.3.345
Marulanda, M., López, A., & Uribe, M. (2012). Genetic Diversity and Transferability of Rubus Microsatellite Markers to South American Rubus Species. The Molecular Basis of Plant Genetic Diversity, 151-164. doi: https://doi.org/10.5772/32838
Murray, M. & Thompson, W. (1980). Rapid isolation of high molecular weight plant DNA. Nucleic Acids Research, 8(19), 4321-4326. doi: https://doi.org/10.1093/nar/8.19.4321
Castillo, N. R. F., Reed, B. M., Graham, J., Fernández-Fernández, F., & Bassil, N. V. (2010). Microsatellite markers for raspberry and blackberry. Journal of the American Society for Horticultural Science, 135(3), 271-278. doi: https://doi.org/10.21273/jashs.135.3.271
Graham, J., Smith, K., MacKenzie, K., Jorgenson, L., Hackett, C., & Powell, W. (2004). The construction of a genetic linkage map of red raspberry (Rubus idaeus subsp. idaeus) based on AFLPs, genomic-SSR and EST-SSR markers. Theoretical and Applied Genetics, 109(4), 740-749. doi: https://doi.org/10.1007/s00122-004-1687-8
Blacket, M. J., Robin, C., Good, R. T., Lee, S. F., & Miller, A. D. (2012). Universal primers for fluorescent labelling of PCR fragments-an efficient and cost-effective approach to genotyping by fluorescence. Molecular Ecology Resources, 12(3), 456-463. doi: https://doi.org/10.1111/j.1755-0998.2011.03104.x
Matlock, B. (2015). Assessment of Nucleic Acid Purity. Recuperado de: https://assets.thermofisher.com/TFS-Assets/CAD/Product-Bulletins/TN52646-E-0215M-NucleicAcid.pdf
Thermo Fisher Scientific Inc. (2009). NanoDrop 2000 / 2000c Spectrophotometer V1.0 User Manual, 97. Recuperado de: https://assets.thermofisher.com/TFS-Assets/CAD/manuals/NanoDrop-2000-User-Manual-EN.pdf
Álvarez, M. (2016). Evaluación de la variabilidad genética en cinco especies de mora (Rubus spp) mediante marcadores microsatélites SSR (Tesis de pregrado). Universidad San Francisco de Quito USFQ, Quito. Recuperado de http://repositorio.usfq.edu.ec/bitstream/23000/5695/1/126382.pdf
Meng, R., & Finn, C. (2002). Determining ploidy level and nuclear DNA content in Rubus by flow cytometry. Journal of the American Society for Horticultural Science, 127(5), 767-775. doi: https://doi.Org/10.21273/jashs.127.5.767
Thompson, M. M. (1995). Chromosome numbers of Rubus species at the national clonal germplasm repository. HortScience, 30(7), 1447-1452. doi: https://doi.org/10.21273/hortsci.30.7.1447
Dossett, M., Bassil, N. V., & Finn, C. E. (2012). High resolution melting detects sequence polymorphism in Rubus occidentalis monomorphic microsatellite markers. Acta Horticulturae, 926, 91-96. doi: https://doi.org/10.17660/actahortic.2012.926.11
Foster, T. M., Bassil, N. V., Dossett, M., Leigh Worthington, M., & Graham, J. (2019). Genetic and genomic resources for Rubus breeding: a roadmap for the future. Horticulture Research, 6(1). doi: https://doi.org/10.1038/s41438-019- 0199-2
Vieira, M. L. C., Santini, L., Diniz, A. L., & Munhoz, C. de F. (2016). Microsatellite markers: What they mean and why they are so useful. Genetics and Molecular Biology, 39(3), 312-328. doi: https://doi.org/10.1590/1678-4685-GMB-2016-0027
Aranguren-Méndez, J. A., Román-Bravo, R., Isea, W., Villasmil, Y., & Jordana, J. (2005). Los microsatélites (STR's), marcadores moleculares de ADN por excelencia para programas de conservación: una revisión Microsatellites (STR’s), ADN Molecular Markers for Excellency for conservation programs: A review. Arch. Latinoam. Prod. Anim., 13(11), 30-42.
Tautz, D., & Schlötterer, C. (1994). Simple sequences. Current Opinion in Genetics and Development, 4(6), 832-837. doi: https://doi.org/10.1016/0959-437X(94)90067-1
Palais, R., & Wittwer, C. T. (2009). Chapter 13 Mathematical Algorithms for High-Resolution DNA Melting Analysis. En M. Johnson, & L. Brand (Eds.), Methods in Enzymology (pp. 323-332). Vol. 454. Elsevier Inc. doi: https://doi.org/10.1016/S0076-6879(08)03813-5
Ng, P. C., & Kirkness, E. F. (2010). Whole Genome Sequencing. Genetic Variation, 215-226. doi: https://doi.org/10.1007/978-1-60327-367-1_12
Ward, J. A., Bhangoo, J., Fernández-Fernández, F., Moore, P., Swanson, J. D., Viola, R., ... Sargent, D. J. (2013). Saturated linkage map construction in Rubus idaeus using genotyping by sequencing and genome-independent imputation. BMC Genomics, 14(1), 1-14. doi: https://doi.org/10.1186/1471-2164-14-2
Ryu, J., Kim, W. J., Im, J., Kim, S. H., Lee, K. S., Jo, H. J., ... Ha, B. K. (2018). Genotyping-by-sequencing based single nucleotide polymorphisms enabled Kompetitive Allele Specific PCR marker development in mutant Rubus genotypes. Electronic Journal ofBiotechnology, 35, 57-62. doi: https://doi.org/10.1016/j.ejbt.2018.08.001
Escobedo López, D., & Núñez Colín, C. (2015). Variabilidad genética de una población segregante de mora mexicana (Morus celtidifolia Kunth) determinada por marcadores ISSR. Acta Agrícola y Pecuaria, 1(3), 93-98.
Amsellem, L., Noyer, J. L., Bourgeois, T. L. E., & Hossaert-Mckey, M. (2000). Comparison of genetic diversity of the invasive weed Rubus alceifolius Poir. (Rosaceae) in its native range and in areas of introduction, using amplified fragment length polymorphism (AFLP) markers. Molecular Ecology, 9(4), 443-455. doi: https://doi.org/10.1046/j.1365-294X.2000.00876.x
Le Bourgeois, T., Baret, S., & de Chenon, R. D. (2011). Biological Control of Rubus alceifolius (Rosaceae) in La Réunion Island (Indian Ocean): From Investigations on the Plant to the Release of the Biological Control Agent Cibdela janthina (Argidae). En Y. Wu, T. Johnson, S. Sing, S. Raghu, G. Wheeler, P. Pratt, K. Warner, T. Center, J. Goolsby, & R. Reardon (Eds.), Proceedings of the XIII International Symposium on Biological Control of Weeds: Session 4 Target and Agent Selection (pp. 153-160), Waikoloa, Hawái, Estados Unidos: U.S. Forest Service.