Artículos
EVALUACIÓN DE LA VARIABILIDAD GENÉTICA DEL TOMATE DE ÁRBOL (Solanum betaceum Cav.) EN LOS CULTIVOS DE TRES PROVINCIAS DEL ECUADOR POR MEDIO DE MARCADORES MICROSATÉLITES.
ACI Avances en Ciencias e Ingenierías
Universidad San Francisco de Quito, Ecuador
ISSN: 1390-5384
ISSN-e: 2528-7788
Periodicidad: Bianual
vol. 1, núm. 1, 2008
Publicación: 01 Abril 2009

Esta obra está bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.
Resumen: El presente trabajo determinó el nivel de diversidad genética existente en varios cultivos de tomate de árbol (Solanum betaceum) de las provincias de Imbabura, Tungurahua y Pichincha, mediante el uso de marcadores microsatélites. Además, se evaluó el nivel de transferibilidad al tomate de árbol, de 53 pares de primers microsatélites desarrollados originalmente para papa (Solanum tuberosum). Se usó 50 accesiones, que abarcaron las seis variedades de tomate de árbol posibles, de acuerdo al tamaño/forma y color del mucílago del fruto. El porcentaje de pares de primers que amplificaron con éxito fue de 28.3 %. Los loci amplificados mostraron mayoritariamente un patrón monomórfico. El porcentaje de primers polimórficos fue de 7.5%. El valor PIC para cada par de primers varió entre 0.077 y 0.514. En solo 5 de los 11 loci utilizados para el análisis, se observaron individuos heterocigotos. El porcentaje de individuos heterocigotos para estos loci varió entre 4 y 98 %. El acervo genético de las accesiones analizadas parece ser estrecho y homogéneo. Se detectaron valores altos de similitud genética entre las accesiones (80 - 100%), usando el coeficiente de Jaccard. El método de agrupamiento UPGMA demostró que las accesiones no se asociaban ni por lugar de origen, ni por variedad. Estos resultados son congruentes con los obtenidos en estudios anteriores. El bajo porcentaje de transferencia de primers (28.3%) indica que las secuencias que flanquean a los microsatélites no se han conservado en el proceso de especiación de la papa y el tomate de árbol. La reducida diversidad genética en los cultivos de tomate de árbol podría explicarse por un proceso relativamente reciente de domesticación de la especie, y por una conjunción de factores geográficos/ecológicos, y fenológicos, que fomentan la homogenización genética entre y dentro de los cultivos.
Palabras clave: Solanum betaceum, tomate de árbol, microsatélites, variabilidad genética.
Introducción
El tomate de árbol (Solanum betaceum) es un frutal de importancia comercial, en Ecuador y en varios países de la Región Andina, de donde es originario [1]. El fruto varía considerablemente en tamaño y aspecto: la piel es usualmente roja, pero oscila entre amarilla hasta púrpura, a veces con rayas longitudinales obscuras; el mucílago que rodea a las semillas puede ser amarillento o morado obscuro; y la forma puede ser desde redonda hasta “puntona” [2]. En el Ecuador, su producción con fines de exportación ha sido afectada tanto por la falta de discriminación varietal en los cultivos, como por el ataque de plagas y enfermedades a las que esta especie es sensible. La aplicación de un programa de fitomejoramiento elevaría el estándar de las plantaciones, lo que a su vez mejoraría la calidad del producto y aliviaría el impacto que las prácticas agrícolas actuales y tradicionales tienen sobre el ambiente [3]. Dentro de tal programa, el análisis molecular es un componente primordial. El conocimiento de la amplitud y la distribución de la diversidad genética dentro de un cultivo resulta invaluable para diversas aplicaciones, incluidas la caracterización molecular de variedades, cultivares e individuos; y la selección de parentales potenciales para proyectos de hibridación e introgresión de genes de interés en el cultivo [4].
Mientras más amplia sea la variabilidad genética de una población, mayores son las opciones para que ésta enfrente con éxito los desafíos bióticos o abióticos en el futuro. La diversidad genética ofrece así, mayores posibilidades de mejorar los cultivos [5].
Los microsatélites (o SSRs) son marcadores moleculares que han sido usados con éxito para varios propósitos desde el fitomejoramiento hasta la evaluación de poblaciones orientada a la conservación [6] en una multitud de especies vegetales, muchas de ellas de importancia comercial [7]. El valor de los microsatélites reside en su condición altamente polimórfica, su naturaleza codominante, su abundancia dentro del genoma, su detección sencilla por PCR, cobertura extensa del genoma y su requerimiento de una pequeña cantidad de ADN para el análisis [6]. El carácter hipervariable de los SSRs permite discriminar eficazmente entre individuos estrechamente relacionados, lo cual es de utilidad para la identificación de cultivares [8].
Sin embargo, el desarrollo de nuevos primers para amplificar secuencias microsatélites siempre involucra un insumo de recursos y tiempo, por lo que en los casos en los que no se puede disponer de primers desarrollados específicamente para la especie de estudio, es frecuente recurrir al uso de primers diseñados a partir de taxa cercanos [9, 10].
La utilidad de la amplificación entre especies responde tanto al porcentaje de primers que pueden ser transferidos, como de la cantidad de polimorfismo que estos muestren, lo cual a su vez varía dependiendo en gran medida del grado en que estén relacionadas las especies involucradas [11].
El presente trabajo establece la variabilidad genética intra- e inter-varietal de los cultivares de tomate de árbol en tres provincias de la Sierra ecuatoriana mediante SSRs y evalúa el grado de transferibilidad al tomate de árbol de 53 pares de primers SSR desarrollados originalmente para papa (Solanum tuberosum).
Materiales y métodos.
Se usó 50 accesiones de tomate de árbol, recogidas en 18 parcelas de cultivo seleccionadas al azar en las provincias de Imbabura, Tungurahua y Pichincha. Se colectó un fruto de cada individuo, al que se lo clasificó, de acuerdo al tamaño y forma de su fruto y al color del mucílago, en una de las siguientes variedades: amarillo común, amarillo gigante, amarillo bola, morado gigante, morado bola y morado común. Las semillas de cada fruto fueron germinadas in vitro en medio MS modificado (30 g de sacarosa y 8 g de agar por litro), para contar con hojas jóvenes para la extracción de ADN, la cual se ejecutó siguiendo el protocolo de CTAB [12]. Para el análisis, se probaron 53 pares de primers desarrollados originalmente para papa (Solanum tuberosum) y proporcionados por el Centro Internacional de la Papa (CIP) de Lima, Perú. Se utilizó geles desnaturalizantes de poliacrilamida al 6% [13] y tinción con plata de acuerdo a Benbouza et al. para evidenciar los resultados [14]. Las bandas en el gel se registraron como presencia (1) o ausencia (0) a partir de lo cual se elaboró una matriz binaria. Estos datos fueron analizados con el programa NTSTSpc 2.11.
Usando el coeficiente de Jaccard, se generó una matriz de similitud entre todas las accesiones, comparadas de dos en dos. Esta matriz fue usada para, mediante el método de agrupamiento UPGMA, producir un dendrograma
El Contenido de Información Polimórfica (PIC por sus siglas en inglés) fue calculado para cada par de primers con la siguiente fórmula: PIC = 1 - Sp;2, donde pi es la frecuencia del alelo i.
Se evaluó el nivel de heterocigosis determinando el porcentaje de individuos heterocigotos para cada locus. De igual manera, se calculó la incidencia de alelos nulos por locus en porcentaje.
Resultados y discusión
Transferibilidad de primers
De los 53 pares de primers probados, 33 (apx. 62%) mostraron productos de amplificación. De entre estos 33, más del 50% presentaba patrones de amplificación inespecífica, en la mayoría de los casos con productos de muy elevado peso molecular. Solamente 15 de los 33 pares de primers (45.5 %; 28.3 % del total de 53) produjeron bandas de micro satélite. Cuatro de estos primers no pudieron ser usados en el análisis definitivo de las muestras porque no se contó con suficiente volumen de los mismos, por lo que solo 11 pares de primers, de entre los 15 originalmente seleccionados, fueron usados en el análisis de las muestras (Tabla 1).
Algunos de los pares de primers amplificaron bandas de micro satélites que estuvieron fuera del rango de peso detectado en papa por el CIP (Tabla 1). En general, los loci amplificados mostraron un patrón monomórfico, constituyendo los individuos heterocigotos la única forma de polimorfismo, de modo que el mayor número de alelos por locus fue de dos. El número de primers polimórficos fue 4 de 53, lo que representa el 7.5%. El valor PIC para cada par de primers varió entre 0.077 y 0.514 (Tabla 1).
| Código Primer | Nombre Primera | Motivo microsatélite | Peso molecular (bp)b | Temperatura annealing (°C)c | Cromosomad | PICe | % Individuos heterocigotos | Incidencia de alelos nulos (%) |
| S036 | STM1053 | (TA)4(ATC)5 | ~ 249 (168-184) | 53(53) | III | 0.211 | - | 12 |
| S089 | STM3012 | (CT)4, (CT)g | ~ 186 (153-213) | 57 (57) | IX | 0.147 | - | 8 |
| S114 | STM5114 | (ACC)7 | a: ~183 b: ~ 293 (288-308) | 57 (57) | II | 0.5 | 98 | 0 |
| S140 | STM5140 | (AAT)9 | a: ~ 177 b: ~ 175 (~200) | 50 (57) | IV | 0.255 | 32 | 0 |
| S182 | STG0004 | (GT)6 | ~ 199 (~200) | 55 (55) | XI | 0.18 | - | 10 |
| S188 | STG0010 | (TG)6 | ~ 173 (150-170) | 54 (55) | III | 0.32 | - | 20 |
| S208 | STG0033 | (CGG)6 | ~ 157 (148-162) | 55 (55) | II | 0.077 | - | 4 |
| S209 | STI0001 | (ATT)n | a: ~ 187 b: ~ 181 (186-208) | 55 (55) | IV | 0.078 | 4 | 2 |
| S221 | STI0023 | (CAG)n | a: ~ 187 b: ~ 184 (130-205) | 50 (60) | X | 0.449 | 68 | 0 |
| S228 | STI0035 | (TTC)n | a y b: < 100 (102-115) | 50 (57) | - | 0.514 | 6 | 38 |
| S231 | STI0038 | (CTG)n | ~ 102 (110-115) | 50 (58) | V | 0.241 | - | 14 |
La incidencia de alelos nulos por locus varió también considerablemente entre un mínimo de 0 y un máximo de 38% de los individuos (Tabla 1).
La utilidad del uso de loci microsatélites entre taxa depende de la conservación de las secuencias a las que se unen los primers y en el mantenimiento, durante largos períodos evolutivos, de secuencias microsatélites lo suficientemente largas como para generar polimorfismo [8]. De manera general, en plantas la amplificación exitosa y útil entre especies parece limitarse a congéneres, reportándose niveles medios a altos (50-100%) de transferibilidad de primers a especies del mismo género, pero no todos ellos son polimórficos (20-100%) [11]. Aun cuando un mismo set de primers se evalúa en especies cercanas del mismo género, tanto el número de primers que amplifican, como el porcentaje de éstos que muestra polimorfismo, varía considerablemente entre dichas especies [9].
De esta manera, en plantas parece ser que la distancia genética y evolutiva existente entre dos especies puede reflejarse en la cantidad de loci que pueden ser amplificados con éxito en las dos especies [10], disminuyendo la claridad y el porcentaje de microsatélites amplificados a medida que aumenta la distancia genética [9], aún entre especies del mismo género [15].
La papa y el tomate de árbol pertenecen al mismo género. Sin embargo, la amplificación aún entre distintos géneros en algunos casos ha arrojado un mayor porcentaje de loci polimórficos que el obtenido en este estudio (7.5%); el porcentaje de loci informativos reportado aquí es inusualmente bajo para primers transferidos dentro del mismo género [9, 10]. Kyndt et al. indican que es de esperar un alto porcentaje de transferibilidad entre especies dentro del mismo género cuando la especiación dentro de ese género ha sido reciente, como sucede con Vasconcellea[9]. En este aspecto, no se cuenta con datos que relacionen a las especies involucradas en este estudio.
Por otro lado, se ha reportado que el tamaño del genoma tiene incidencia en la eficiencia de la amplificación de los loci microsatélites. La tendencia es que mientras más grande sea el genoma de una especie, mayor es la proporción de primers que no logran producir una amplificación exitosa del ADN [16]. Los genomas del tomate de árbol y de sus especies relacionadas sobresalen por su tamaño: incluso el menor de ellos tiene más ADN que otro taxón de igual número cromosómico (24) dentro de las solanáceas [17].
El número de loci finalmente analizados en este estudio
(11) es superior al promedio (8) que se maneja comúnmente en los estudios de variabilidad genética intraespecífica en plantas usando microsatélites [18]. Sin embargo, es recomendable incrementar el número de primers, para incluir al menos un locus por cromosoma y así tener una mejor cobertura del genoma [19].
| Criterio de agrupación | Prom. | SD | Max. | Min. |
| Lugar de origen | ||||
| Imbabura | 0,89 | 0,098 | 1,0 | 0,64 |
| Tungurahua | 0,87 | 0,090 | 1,0 | 0,64 |
| Pichincha | 0,83 | 0,111 | 1,0 | 0,50 |
| Variedades | ||||
| Amarillos | 0,86 | 0,089 | 1,0 | 0,60 |
| Morados | 0,84 | 0,114 | 1,0 | 0,50 |
| Comunes | 0,83 | 0,109 | 1,0 | 0,50 |
| Gigantes | 0,89 | 0,088 | 1,0 | 0,62 |
| Bola | 0,87 | 0,098 | 1,0 | 0,60 |
Tabla2. Valores de similitud genética (coeficiente de Jaccard) de 50 accesiones de tomate de árbol, agrupados por variedades y lugar de origen.
Variabilidad genética
El acervo genético de las accesiones analizadas parece ser estrecho y homogéneo. Se detectaron valores altos de similitud genética entre las accesiones (80 - 100%) (Figura 1). El valor promedio de similitud genética entre las 50 accesiones analizadas con los SSRs fue de 0.85 ± 0.10. Aún dentro de cada provincia y dentro de cada variedad, el promedio de similitud genética estuvo por encima del 80% (Tabla 2).
El dendrograma (Figura 1) mostró 23 genotipos distintos entre las 50 accesiones. En él no se observó un patrón de agrupamiento que respondiera ni al origen de las accesiones ni a ninguna característica varietal. Estos resultados son congruentes con los obtenidos en otros estudios, en los que se usaron AFLPs y microsatélites para determinar la diversidad genética de S. betaceum en el país [20, 21].
Se suele asociar un patrón de diversidad genética reducida con algún suceso debido al cual el número de individuos de una población disminuyó, reduciendo así también la variedad de genotipos o alelos presentes en esa población. Esta ha sido la regla para las especies vegetales que han sido sujeto de domesticación [5, 22]. La reducción de diversidad genética en las variedades de los cultivos se ha observado en casos como el trigo (Triticum aestivum), debido a una domesticación relativamente reciente [22].
Aunque no queda claro todavía dónde ni cuándo se empezó a cultivar el tomate de árbol en Sudamérica, parece ser que su domesticación es más bien reciente [1]. Reducida diversidad genética ha sido reportada también para sandía [23], presumiblemente debido al bajo número de plantas usadas en los primeros estadíos de la domesticación de este cultivo fuera de su área de origen [24].
El factor geográfico puede jugar también un papel determinante en los patrones de diversidad genética Para ciertas especies vegetales, el aislamiento geográfico producido por la fragmentación del hábitat ha propiciado la diferenciación genética entre poblaciones, diferenciación que ha sido reforzada por la floración asincrónica entre esas poblaciones [25].
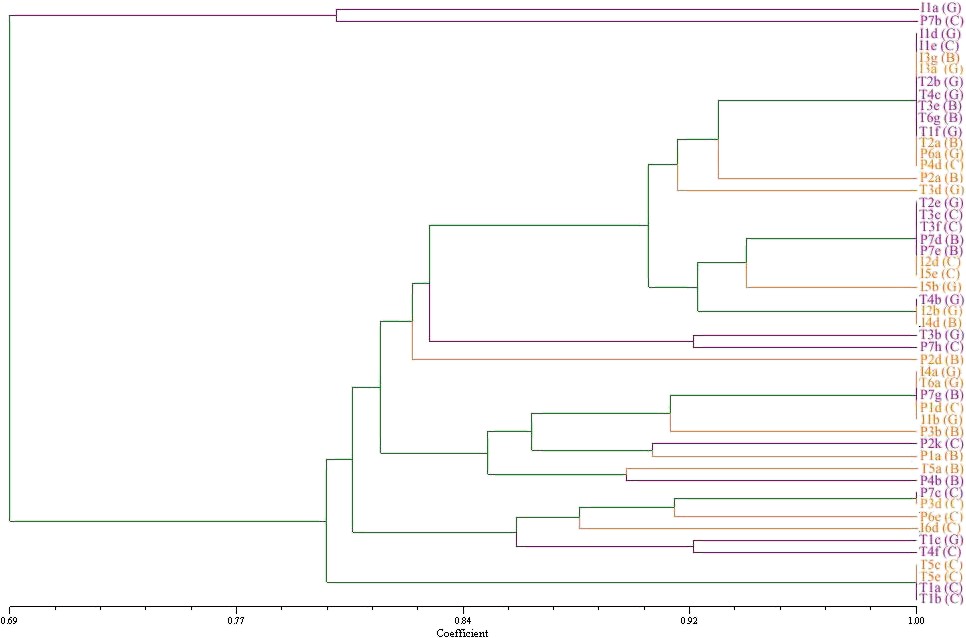
Figura 1
Dendrograma generado por el método UPGMA usando el coeficiente de similitud de Jaccard a partir de los patrones de bandas generadas por 11 pares de primers de SSRs en 50 accesiones de tomate de árbol La primera letra de cada accesión denota su origen I para Imbabura T para Tungurahua y P para Pichincha El color de cada accesión corresponde a las variedades Morado o Amarilla y las letras entre paréntesis corresponden a las variedades Gigante G Común C o Bola B El eje horizontal indica el valor de similitud entre las accesiones
En este sentido, las características geográficas donde se da el cultivo del tomate de árbol y la fenología de la planta parecen contribuir a que todas las poblaciones de este cultivo en el Ecuador sean homogéneas entre sí. El tomate de árbol en nuestro país se siembra en un continuo en todas las provincias de la Sierra, de modo que quizás la división en poblaciones por provincia o localidad sea un artificio porque en realidad las plantaciones de todo el Ecuador podrían constituir genéticamente una sola población. Las condiciones climáticas en las que se produce este cultivo no difieren mayormente entre sí, por lo que no se ha producido una adaptación local a un ambiente específico. Esto, sumado a que en el tomate de árbol predomina la exogamia a través de la polinización mediada por insectos [26], contribuiría a la homogeneización del acervo genético dentro del tomate de árbol cultivado.
A pesar de observarse una limitada variabilidad genética en el tomate de árbol cultivado, existe una gran variación morfológica en el fruto. Un fenómeno análogo detectado entre cultivares de sandía [23] y en árboles del género Panax[27] parece deberse a que la mayoría de las características que distinguen a los cultivares son caracteres cualitativos determinados por una o pocas mutaciones que no pudieron ser detectadas por el método empleado para el análisis [23,27]. Hasta qué punto estos escenarios se corresponden con la situación del tomate de árbol no ha sido determinado.
Los resultados presentados revelan una reducida diversidad genética en las plantaciones de S. betaceum en el Ecuador y constituyen un primer paso para determinar futuras acciones en el mejoramiento de este cultivo.
Agradecimientos
Quiero reconocer el apoyo brindado por el CONESUP a través del financiamiento brindado para la realización de este proyecto.
Referencias bibliográficas
1. Bohs, L. 1989. “Ethnobotany of the Genus Cyphomandra (Solanaceae)”. Economic Botany, 43(2): 143-163.
2. Albornoz, G. 1992. El tomate de árbol (Cyphomandra betacea Sendt.) en el Ecuador. Universidad Central del Ecuador-Fundagro, Quito.
3. Morales, J.O. 2001. “Diagnóstico agro-socio- económico del cultivo de tomate de árbol (Cyphomandra betacea Sendt.) en cuatro provincias de la sierra ecuatoriana (Imbabura, Pichincha, Tungurahua y Azuay)”. Tesis de grado previa a la obtención de título de Ingeniero Agrónomo de la Universidad Central del Ecuador, Quito.
4. Mohammadi, S.A. & Prasanna, B.M. 2003. “Analysis of Genetic Diversity in Crop Plants - Salient Statistical Tools and Considerations”. Crop Science: 43, 1235 -1248.
5. Tanksley, S.D.; Mccouch, S.R. 1997. “Seed Banks and Molecular Maps: Unlocking Genetic Potential from the Wild”. Science 277: 1063-1066.
6. Nuez, F.; Carrillo, J.M. (Eds.). 2000. Los marcadores genéticos en la mejora vegetal. Editorial Universidad Politécnica de Valencia. Valencia, España.
7. Azofeifa-Delgado, A. 2006. “Revisión Bibliográfica: Uso de marcadores moleculares en plantas; aplicaciones en frutales del trópico”. Agronomía Mesoamericana 17(2): 221-242.
8. Powell, W.; Machray, G.C.; Provan, J. 1996. “Polymorphism revealed by simple sequence repeats.” Trends in Plant Science 1(7): 215-222.
9. Kyndt, T., Van Droogenbroeck, B., Haegeman, A., Roldán-Ruiz, I. & Gheysen, G. 2006. “Cross-species microsatellite amplification in Vasconcellea and related genera and their use in germplasm classification.” Genome 49: 786-798.
10. Shepherd, M.; Cross, M.; Maguire, T.L.; Dieters, M.J.; Williams, C.G.; Henry, R.J. 2002. “Transpecific microsatellites for hard pines”. Theor Appl Genet 104: 819-827.
11. Peakall, R.; Gilmore, S.; Keys, W.; Morgante, M.; Rafalski, A. 1998. “Cross-species amplification of soybean (Glycine max) Simple Sequence Repeats (SSRs) within the genus and other legume genera: implications for the transferability of SSRs in plants.” Mol. Biol Evol 15(10): 1275-1287.
12. Saghai Maroof, M.A.; Solima, K.M.; Jorgenson, R.A.; Allard, R.W. 1984. “Ribosomal DNA spacer- length polymorphisms in barley: Mendelian inheritance, chromosomal location, and population dynamics.” Proc. Nac. Acad. Sci. USA 81: 80148018.
13. CIP - Centro Internacional De La Papa. 2000. Molecular Biology Laboratory Protocols: Plant Genotyping. Ma. del R. Herrera; M. Ghislain; D. Zhang (Eds.), Crop Improvement and Genetic Resources Department Training Manual. Tercera edición, Lima, Perú.
14. Benbouza, H.; Jacquemin, J.M.; Baudoin, J.P.; Mergeai, G. 2006. “Optimization of a reliable, fast, cheap and sensitive silver staining method to detect SSR markers in polyacrylamide gels.” Biotechnol. Agron. Soc. Environ. 10 (2): 77-81.
15. Escribano, P.; Viruel, M.A.; Hormaza, J.I. 2004. “Characterization and cross-species amplification of microsatellite markers in cherimoya (Annona cherimola Mill., Annonaceae)”, Primer Note. Molecular Ecology Notes 4: 746-748.
16. Garner, T.W.J. 2002. “Genome size and microsatellites: the effect of nuclear size on amplification potential.” Genome 45: 212-215.
17. Pringle, G.J.; Murray, B.G. 1991. “Karyotype Diversity and Nuclear DNA Variation in Cyphomandra” en Solanaceae III: Taxonomy, Chemistry, Evolution; ed. Hawkes, Lester, Nee & Estrada; Royal Botanic Gardens Kew and Linnean Society of London.
18. Nybom, H. 2004. “Comparison of different nuclear DNA markers for estimating intraspecific genetic diversity in plants.” Molecular Ecology 13: 11431155.
19. Ghislain, M.; Spooner, D.M.; Rodríguez, F.; Villamón, F.; Núñez, J.; Vásquez, C.; Waugh, R.; Bonierbale, M. 2004. “Selection of highly informative and user-friendly microsatellites (SSRs) for genotyping of cultivated potato.” Theor Appl Genet 108: 881-890.
20. Tapia, C.; Zambrano, E.; Morillo, E. 2006. “Tomate de árbol (Cyphomandra betacea Sendt.), frutal promisorio para la diversificación del agro andino.” Informe de Proyecto ejecutado por INIAP/DENAREF.
21. Ordóñez, S. 2007. “Diferenciación de variedades en cultivos de tomate de árbol, Solanum betaceum, mediante la técnica molecular de AFLP.” Tesis degrado, presentada como requisito para la obtención de título de B.S. en Biotecnología de la Universidad San Francisco de Quito. Quito, Ecuador.
22. Reif, J.C.; Zhang, P.; Dreisigacker, S.; Warburton, M.L.; Van Ginkel, M.; Hoisington, D.; Bohn, M.; Melchinger, A.E. 2005. “Wheat Genetic Diversity Trends During Domestication and Breeding.” Theor Appl Genet 110: 859-864.
23. Levi, A.; Thomas, C.E.; Whener, T.C.; Zhang, X. 2001. “Low genetic diversity indicates the need to broaden the genetic base of cultivated watermelon.” HortSience 36 (6): 1096-1101.
24. Navot, N.; Zamir, D. 1987. “Isozyme and seed protein phylogeny of the genus Citrullus (Cucurbitaceae).” Plant Syst. Evol. 156: 61-67.
25. González-Astorga, J.; Castillo-Campos, G. 2004. “Genetic Variability of the Narrow Endemic Tree Antirhea aromatica Castillo-Campos & Lorence, (Rubiaceae, Guettardeae) in a Tropical Forest of Mexico.” Annals of Botany 93: 521-528.
26. Lewis, D.H.; Considine, J.A. 1999. “Pollination and fruit set in the tamarillo (Cyphomandra betacea (Cav.) Sendt.) 1. Floral biology.” New Zealand Journal of Crop and Horticultural Science 27: 101112.
Zhou, S.L.; Xiong, G.M.; Li, Z.Y.; Wen, J. 2005. “Loss of genetic diversity of domesticated Panax notoginseng F H Chen as evidenced by ITS sequence and AFLP polymorphism: a comparative study with P. stipuleanatus. H T Tsai et K M Feng.” Journal of Integrative Plant Biology 47(1): 107-115.

