



Artículos
DIFERENCIACIÓN MOLECULAR EN VARIEDADES CULTIVADAS DE TOMATE DE ÁRBOL (Solanum betaceum), MEDIANTE LA TÉCNICA MOLECULAR DE AFLP
ACI Avances en Ciencias e Ingenierías
Universidad San Francisco de Quito, Ecuador
ISSN: 1390-5384
ISSN-e: 2528-7788
Periodicidad: Bianual
vol. 1, núm. 1, 2008
Publicación: 01 Abril 2009

Resumen: Esta investigación tuvo como objetivo determinar molecularmente el nivel de variabilidad genética intra e inter varietal existente en los cultivos de Solanum betaceum en tres provincias del Ecuador, Imbabura, Pichincha y Tungurahua y, basado en esto, identificar las seis variedades reconocidas desde el punto de vista morfológico (amarillo puntón, amarillo gigante, amarillo bola, morado gigante, morado bola y morado común). Se incluyó de cuatro a cinco individuos por variedad, para un total de 26 accesiones objeto del estudio. La técnica usada fue la amplificación al azar de fragmentos polimórficos de ADN, AFLP. Se seleccionó 8 combinaciones de primers que se consideraron como las más informativas por el número y definición de bandas en el gel y el nivel de polimorfismo develado. Se encontró variabilidad limitada entre accesiones, teniendo un máximo del 18% de distancia entre genotipos del tipo amarillo con respecto a los del tipo morado, pero no fue posible distinguirlas con respecto a la forma del fruto ni a la provincia de origen. Las combinaciones de primers usadas, detectaron un 60% de polimorfismo entre accesiones y permitieron establecer que los individuos muestreados no representaban genotipos completamente diferenciables debido quizá a su elevado nivel de cruzamiento intervarietal. En futuros estudios sería necesario incluir individuos de poblaciones silvestres que puedan aportar información sobre los orígenes de la variabilidad existente en el cultivo de tomate de árbol del Ecuador.
Palabras clave: AFLP, Polimorfísmo, Solanum betaceum..
Introducción
Solanum betaceum o tomate de árbol es un arbusto endémico de la región oriental de los Andes [1]. Se caracteriza por su fruto ovoide o redondeado, dependiendo de la variedad. Su mesocarpio es de color amarillo, morado o tomate, con cáscara lisa y gruesa variando su color de rojo a amarillo [2]. Albornoz y Cadena, por estudios fenotípicos reconocen que en Ecuador existen 5 variedades. Dos variedades similares a las que debieron ser las primeras plantas de tomate de árbol domesticadas, (amarilla y negra), una tercera variedad “Criollo”, dividida en dos subvariedades, (puntón y redondo), y una quinta variedad roja [3,4]. En esta investigación se usa las variedades que distingue el agricultor y que resultan de la combinación de dos caracteres del fruto: color (amarillo o morado) y forma (puntón, gigante o bola), dando un total de 6 posibles variedades (Figura 1).

El tomate de árbol es un cultivo económicamente importante en los países andinos como Ecuador, Colombia y Perú. Ecuador empezó a exportar esta fruta en la década de los 80`s pero hasta ahora no ha logrado ampliar considerablemente el mercado de exportación. Una razón es la dificultad de producir frutos homogéneos en forma y color. [3,5]
Debido a la poca información existente sobre la variabilidad genética de especies endémicas, basada en estudios moleculares, esta investigación tuvo como objetivo principal iniciar el estudio de la variabilidad inter e intra varietal del tomate de árbol en los actuales cultivos ecuatorianos, centrándose en las provincias de Pichincha, Tunguragua e Imbabura. Conociendo la variabilidad genética de la especie a futuro se podrá desarrollar estrategias de conservación para evitar una posible erosión genética, encontrar variedades resistentes a plagas, mejorar la selección en cultivos y el rendimiento de los mismos y, en el aspecto económico, ampliar el mercado de exportación.
En este estudio se utilizó la técnica AFLP que es una de las más efectivas para la obtención de un gran número de marcadores de loci independientes no específicos. Comparado con otras técnicas, los marcadores AFLPs presentan algunas ventajas, una de estas es la obtención de mayor información sobre variaciones dentro de una especie [6]. En investigaciones como la actual, con una especie que no ha sido estudiada anteriormente a nivel molecular, y donde se conoce que la variabilidad es escasa, los marcadores AFLP han dado buenos resultados
Materiales y métodos
Material vegetal:
Se recolectó frutos maduros de las seis variedades de tomate de árbol (amarillo puntón, amarillo bola, morado gigante, morado puntón, morado bola, y amarillo bola), en las provincias de Pichincha, Tungurahua e Imbabura.
Se extrajo las semillas separándolas por variedad y por individuo, se lavaron en agua corriente y se las dejó secar por 24 horas. El proceso de esterilización comenzó sumergiendo las semillas por 5 minutos en etanol 70%, y luego fueron lavadas en agua destilada estéril. Posteriormente se sumergieron, durante 20 minutos, en una solución de hipoclorito de sodio al 2.5% a la que se adicionó cuatro gotas de Tween 20. Finalmente, las semillas fueron lavadas cuatro veces con agua destilada estéril.
Se sembró las semillas en medio MS con 30 g/L de sacarosa y 7 g/L de agar a pH 5.8 [9]. El número de semillas por frasco fue de 6 a 7. Durante el proceso de germinación y crecimiento, las semillas estuvieron en un cuarto de cultivo con un fotoperiodo de 16 horas luz/8 horas oscuridad, a 22°C de temperatura. Cuando las plantas tuvieron un tamaño de más o menos 4 cm. se las subcultivó en medio MS [9].
Extracción y cuantificación de DNA:
De las plantas crecidas in vitro se maceró dos hojas por planta (total 26 plantas) en un mortero con 800 µl de buffer de extracción CTAB hasta obtener una solución homogénea la cual se transfirió a un tubo eppendorf de 1.5ml, adicionando 8 µl de -mercapto etanol. Seguidamente, se mantuvo a 62°C por una hora, agitando cada 15 minutos.
Se adicionó 500 µl de cloroformo/ alcohol isoamílico 25:1 y se agitó hasta obtener una mezcla homogénea, la misma que se dejó en reposo por 20 minutos. Luego se centrifugó los tubos a 13.000 rpm por 20 minutos. El sobrenadante se transfirió a otro tubo eppendorf donde se adicionó un volumen de isopropanol agitando hasta que los hilos de ADN fueron visibles. Por último, el pellet de ADN fue separado y transferido a otro tubo eppendorf, donde se lavó tres veces con etanol al 76%/ 0.2M de Acetato de Sodio, 76% alcohol/10mM Acetato de amonio y alcohol al 70% respectivamente. El pellet se secó a temperatura ambiente y se resuspendió en 70 µl de TE estéril [10]. El ADN fue cuantificado en un espectofotómetro Pharmacia Biotech Ultraespec 2000 y se diluyó cada muestra para obtener una concentración final de 100ng/ µl.
Amplificación AFLPs:
Se utilizó el protocolo del manual de AFLP de Invitrogen, que consta de las siguientes etapas :
Restricción: Se realizó adicionando 2 µl de EcoR1/Mse1 a 5ul de la muestra diluida de ADN, 5 µl del buffer de reacción y 13 µl de agua destilada estéril, en un tubo eppendorf de 1.5ml. Se centrifugó por pocos segundos y se llevó a 37°C por dos horas. Luego de lo cual se mantuvo a 70°C por 15 min y finalmente se centrifugó antes de guardar los tubos a -20°C.
Ligación de los adaptadores: A la muestra de ADN previamente cortada con las enzimas EcoR1 y Mse1, se le adicionó 24 µl de la solución de adaptadores proveniente del Kit de AFLPs y 1µl de T4 ADN ligasa, centrifugando por pocos segundos. Como paso final, la mezcla se incubó a temperatura ambiente por 1h y se guardó a -20°C.
Preamplificación: En un tubo de microcentrífuga de 0.2 ml. se adicionó 5 µl de la muestra de ADN previamente ligada con adaptadores, 40 µl de la solución Preamp primer Mix1, 5 µl del buffer 10x para PCR y 1 µl de Taq polimerasa. El ciclaje de la PCR se realizó según el manual del Kit de AFLP de Invitrogen.
Amplificación selectiva: se usó ocho combinaciones de primers (Tabla 1), junto con las 26 muestras de ADN anteriormente preamplificadas. La mezcla de reacción por cada muestra contenía 5 µl de la solución con los primers, 10 µl de la solución de polimerasa y 5 µl de ADN de la muestra específica preamplificada. El ciclo del PCR fue igual al descrito en el manual del Kit de AFLP de Invitrogen.De la amplificación selectiva solo 24 muestras pudieron ser analizadas, de las cuales cinco correspondieron a la variedad amarillo gigante, cinco a morado gigante, cinco al amarillo común, cinco a las variedades Bola, y cuatro a la variedad morado común o puntón
Electroforesis:
La comprobación de los resultados obtenidos en cada etapa del proceso de AFLPs, como también, para la elección de los pares de primers a utilizarse en la amplificación selectiva, se realizó en gel de agarosa al 1%, adicionando 5 µl de bromuro de etidio por cada 100 ml de gel. El gel corrió durante dos horas a 80 V.
Los productos de la amplificación selectiva de cada una de las muestras con los diferentes pares de primers se analizaron en geles denaturalizantes de poliacrilamida 6% [11]. Cada posillo del gel llevó 5µl de la muestra respectiva y se utilizó 1µl de ADN ladder 100 bp de Invitrogen en una relación de 1:3 con el buffer de carga, se corrió a 85 a 100 watts por 2h y 30', de 45 a 50°C.
Tinción con plata: Para la tinción del gel de poliacrilamida, se usó el protocolo de Benbouza H. [12]. Selección de primers: se probaron 44 combinaciones de primers sugeridas en el manual del Kit AFLP de Invitrogen, utilizando una muestra de ADN de tomate de árbol del tipo “amarillo”.
Análisis de datos: Las bandas observadas mediante tinción con plata en los geles de poliacrilamida se transformaron a datos binarios y se construyeron matrices básicas de datos (MDBs). Las bandas con buena resolución se registraron como (1) presencia o (0) ausencia y esos datos fueron ingresados en una tabla de Excel. Para la transformación de las matrices de datos a matrices de similitud se utilizó el programa NTSYCpc 2.11. Los coeficientes de asociación elegidos para el análisis de cada combinación de primers fueron Dice y Nei.
De las matrices de similitud obtenidas, con el coeficiente de Dice, se construyeron árboles fenéticos usando la opción “Clustering” de NTSYs y dentro de ésta la opción “SAHN” (Sequential Agglomerative Hierarchical Nesting, por sus siglas en inglés), y UPGMA (Unweighted Pair Group Method, Arithmetic averaging, por sus siglas en inglés). Con la matriz de distancias generada con el coeficiente de Nei se utilizó la opción “Neighbor joining” para el agrupamiento de las muestras.
La validez de los árboles fenéticos se comprobó mediante un análisis del coeficiente de correlación co-fenético, realizado también con el programa NTSYSpc 2.11
Resultados y discusión
Selección de primers:
Selección de primers: Los pares de primers puestos a prueba, en su mayoría, mostraron productos de amplificación del ADN; por su definición y claridad se usaron las siguientes combinaciones; 18 (M-CAA/E- AAG), 26 (M-CAC/E-ACA), 6 (M-CTG/E-ACA), 7 (M-CAA/E-ACG), 8 (M-CTC/E-ACG), 43 (M-CTG/E-ACG), 46 (M-CAC/E-ACT), y 5 (M-CAT/E-ACT).
Variabilidad:
Variabilidad: Con los ocho pares de primers eleccionados para la amplificación selectiva se obtuvo un total de 183 marcadores, siendo 110 polimórficos (60%) (Tabla 1). Este porcentaje de polimorfismo indica que hay cierto grado de variabilidad genética, entre los genotipos analizados, que podría ser utilizada con fines de mejoramiento. [13]
Los pesos de las bandas encontradas variaron entre 800 a 100 pb. Las bandas utilizadas para el análisis fueron las que presentaron mayor definición en el gel de acrilamida; y fueron las bandas comprendidas entre los 200 pb.y los 600 pb.
El número de bandas generadas por cada combinación de primers fue de 8 a 46. Esto tiene mucho que ver con la secuencia de los tres últimos nucleótidos en los primers selectivos. La cantidad de Citocinas y Guaninas, vuelve al primer más selectivo y hace más difícil la unión al ADN que los nucleótidos A, T. Esto se debe a que las regiones con más pares C-G son más compactas, haciendo más difícil la unión del primer.
Otra de las razones es que, en las plantas la concentración de pares A-T es mucho mayor a la de C-G, por lo que los pares de primers con mayor cantidad de A-T tendrán mayor oportunidad de unirse al DNA, lo que concuerda con los resultados obtenidos. [14]
| Número | Primers selectivos | Marcadores obtenidos | Marcadores polimóricos | Polimorismo por combinación de primers(%) |
| 18 | M-CAA E-AAG | 34 | 19 | 53.4% |
| 26 | M-CAC E-ACA | 46 | 36 | 78.3% |
| 6 | M-CTG E-ACA | 21 | 11 | 52.4% |
| 7 | M-CAA E-ACG | 18 | 9 | 50% |
| 8 | M-CTC E-ACG | 21 | 14 | 66.7% |
| 43 | M-CTG E-ACG | 8 | 8 | 100% |
| 46 | M-CAC E-ACT | 25 | 10 | 40% |
| 5 | M-CAT E-ACT | 10 | 3 | 30% |
La variabilidad observada entre genotipos no fue mayor al 20%, tanto entre variedades, como a nivel intravarietal. Esto puede deberse a que cada genotipo provino de una semilla distinta, aumentando así las diferencias entre individuos. Otra razón puede ser que debido a la mezcla de variedades en los campos de cultivo y a un cierto nivel de alogamia, es posible que estén ocurriendo cruzamientos espontáneos entre variedades.
El nivel de variabilidad encontrado permitió diferenciar los genotipos solo en relación al color del fruto, más no con respecto a la forma del fruto ni al origen del mismo.
Análisis de similitud y agrupamiento:
Se analizaron por separado los datos generados por cada par de primers y luego se combinaron aquellos cuyos resultados eran más congruentes con las características morfológicas; con ellos se construyó dos fenogramas finales utilizando el coeficiente de Dice y Nei respectivamente.
Las combinaciones de primers que generaron la mayor cantidad de datos y lograron agrupar los diferentes genotipos por color de fruto fueron: 8 (M-CTC/E- ACG), 18 (M-CAA/E-AAG), 26 (M-CAC/E-ACA), y 46 (M-CAC/E-ACT).
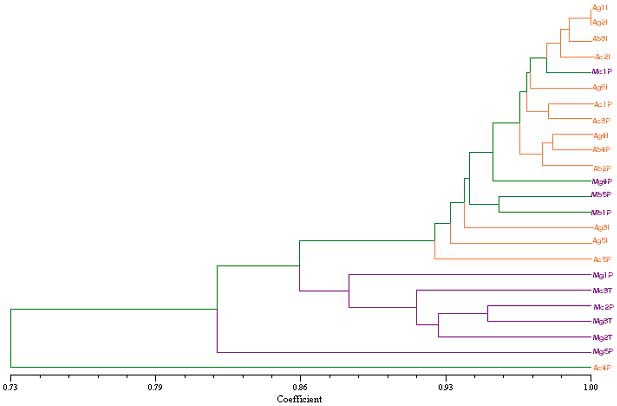
El fenograma UPGMA (Figura 2) construido a partir de la matriz generada con el algoritmo de Dice, distingue dos grupos, uno incluye la mayoría de genotipos del tipo morado y el otro incluye a la mayoría del tipo amarillo, con un coeficiente de similitud de 0.86 entre ellos. Como excepción, dentro del grupo del tipo amarillo, se encuentran dos genotipos, B5 y B4, de la variedad Bola morado, según la clasificación que da el agricultor. Otras excepciones son la agrupación de los genotipos Mc1P y Mg4P del tipo morado, que se acerca más a los genotipos del tipo amarillo y la separación del genotipo Ac4P con un coeficiente de 0.73 con respecto a las demás muestras.
El coeficiente de similitud (0.86) obtenido en el fenograma final (con el coeficiente de Dice), indica un 14% de diferencia entre el grupo que contiene a los genotipos amarillos y el grupo que contiene a los genotipos morados. Esto talvez no sea suficiente como para determinar una separación a nivel de variedades, aunque el porcentaje de distancia específico requerido para determinar una variedad no está definido.[15]
El fenograma también sugiere que las muestras que se analizaron representaban a genotipos producto de la mezcla entre las diferentes “variedades” en el área de cultivo. Esta observación también sustentaría lo plan- teado por Albornoz. [3]
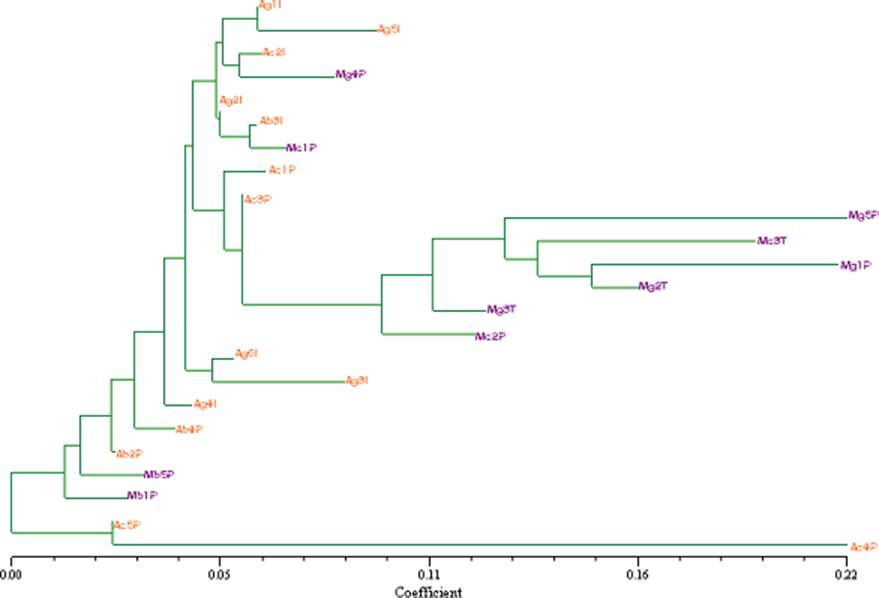
En el fenograma obtenido con la función “Neighbor joining” (Figura 3), se puede ver también la diferencia entre los genotipos del tipo morado y los del tipo amarillo. El grupo que contiene a las muestras del tipo amarillo se encuentra “dividido” en dos, ya que el grupo morado, aunque más distante del resto de muestras (0.11 de distancia), se ubica en el centro.
El genotipo Ac4P parece ser distinto a todas las demás muestras, con una distancia de más o menos 0.20 del genotipo más cercano, que es Ac5P.
El valor co-fenético del fenograma final se obtuvo como se describe en la metodología. El valor r= 0.91 obtenido indica que el grado de distorsión del fenograma con res- pecto a la matriz de similitud de la cual fue obtenido es de 0.09 y se acerca al ideal [16].
En resumen, el número de muestras y combinación de primers utilizados fueron suficientes para separar a los genotipos por el color del fruto (amarillo o morado). Comparado con otros estudios que tratan sobre variabilidad intravarietal, el número de pares de primers y el número de muestras utilizados es similar [13, 17]. Aunque las agrupaciones de las muestras de tomate de árbol obtenidas en los fenogramas no diferencian a los genotipos por la forma del fruto, sino solo por color, es posible que aumentando la combinación de primers se puedan encontrar marcadores ligados a las características morfológicas que son usadas por Albornoz para definir las variedades.[3].
Con esta investigación se puede concluir que las plantaciones de Solanum betaceum en la sierra ecua- toriana están conformados por cultivos heterogéneos. Posiblemente la falta de selección y separación entre los individuos produjo que el actual cultivo no tenga genotipos completamente diferenciables, sino una mezcla, que puede haber sido el producto del cruce entre posibles variedades iniciales.Estudios posteriores deberán abarcar un análisis más amplio de marcadores moleculares y la inclusión de accesiones silvestres para obtener información más completa sobre el origen de la variabilidad presente en el tomate de árbol cultivado en el Ecuador.
Agradecimientos
Al grupo de Investigación del Laboratorio de Agro- Biotecnología de la Universidad San Francisco: en especial a Andrea Arias y Nicolás Peñafiel, por el apoyo con experiencia durante toda la investigación hasta su culminación. Esta investigación fue financiada por el convenio USFQ-CONESUP dentro del marco del proyecto: “Estrategias biotecnológicas para la determinación de la variabilidad genética y la búsqueda de resistencia al nemátodo agallador en tomate de árbol”
Referencias bibliográficas
1. LEBN, Juan, 1996, “A guide for the cultivation of tomarillo”, INIAP-COTESU
2. Aristizábal J.C. “Efecto de las Podas, Distancias de Siembra y Sistemas de Manejo Sobre el Crecimiento y Producción del Tomate de Árbol(Solanum betaceum Sendt.)”. Frutales 19 (1998).
3. Albornoz G. “El tomate de árbol, Determinación de las características varietales”. Universidad Central del Ecuador (1992).
4. Cadena E. “Estudio de Prefactibilidad para el Tomate de Árbol”. SICA, Ministerio de Agricultura y Ganaderia del Ecuador. 13 Enero 2007.<http://www. sica.gov.ec /agronegocios>
5. Tápia Cesar, Eddie Zambrano, Eduardo Morillo. “Tomate de árbol ( Ciphomandra betacea sendt.) frutal promisiorio para la diversificación del agro andino”. INIAP. 2006
6. Manubens A., Sergio Lobos, Yael Jadue, Manuel Toro, Rosa Messina, Manuel Lladser1 y Daniela Seelenfreund. “DNA Isolation and AFLP Fingerprinting of Nectarine and Peach Varieties (Prunus persica)”. Plant Molecular Biology Reporter 17 (1999) 255–267.
7. INCIRLI A., Hatice BILGI., y M. S. AKKAYA. “Assessment of Polymorphic AFLP Markers in Triticum durum and Aegilops sp.” Middle East Technical University (2000).
8. Matthew D., Perry Michael R., Davey J., Brian Power, Kenneth C,. Lowe H. Frances J. Bligh Paul S., Roach and Chris Jones.“DNA Isolation and AFLPTM Genetic Fingerprinting of Theobroma cacao (L.)”.Plant Molecular Biology Reporter 16: 49–59, (1998)
9. Murashige, T y F. Skoog. “.A revised medium for rapid growth and bioassays with tobacco tissue cultures”. Physiol. Plant 15 (1962): 473-497.
10. Shagai-Maroof M.A., Soliman K.M. Jorgensen R.A. y Allard R.W., 1984. Ribosomal DNA spacer-length polymorphisms in barley: Mendelian inheritance, chromosomal locations, and populations dynamics. Proc Natl Acad Sci USA 81: 8014-8018.
11. Sambrook, J., Fritsch, E. F., Maniatis, T. (1989) Molecular Cloning: A Laboratory Manual , 2nd Ed. , Cold Spring Harbor Laboratory, Cold Spring Harbor, NY.
12. Benbouza, H., Jacquemin, J.M., Baudoin, J.P. & Mergeai, G. 2006. “Optimization of a reliable, fast, cheap and sensitive silver staining method to detect SSR markers in polyacrylamide gels.” Biotechnol. Agron. Soc. Environ. 10 (2): 77-81.
13. Manubens A., Sergio Lobos, Yael Jadue, Manuel Toro, Rosa Messina, Manuel Lladser1 y Daniela Seelenfreund. “DNA Isolation and AFLP Fingerprinting of Nectarine and Peach Varieties (Prunus persica)”. Plant Molecular Biology Reporter 17 (1999) 255–267.
14. S. Coulibaly · R.S. Pasquet · R. Papa · P. Gepts AFLP analysis of the phenetic organization and genetic diversity of Vigna unguiculata L. Walp. reveals extensive gene :fow between wild and domesticated types. Theor Appl Genet (2002) 104:358–366.
15. Rohlf F.J. “Guía para el uso del programa NTSYS- pc” Introducción a la Taxonomía (1998).
16. Pringle G. J, Brian G. Murray. “ Karyotype Diversity and Nuclear DNA Variation in Ciphomandra”. Solanaceae III. Royal Botanic Gardens Kew and Linnean Society of London (1991).
Van Droogenbroeck B., P. Breyne, P. Goetghebeur, E. Romeijn-Peeters, T. Kyndt, G. Gheysen. “AFLP analysis of genetic relationships among papaya and its wild relatives (Caricaceae) from Ecuador”. Theor Appl Genet (2002) 105:289–297.

